国家生物信息中心揭示转录因子局部或远程互作的新模式
由架构型(architectural)或组织特异型(tissue-specific)转录因子介导的顺式调控元件之间的染色质相互作用,是发生在细胞核中的许多重要生命活动的基础,如转录、DNA复制和DNA损伤修复等。目前,基于邻近连接的Hi-C及各种衍生方法是研究染色质相互作用的主流手段。但是这些方法通常使用与染色质相互作用本身功能无关的工具酶对基因组进行片段化,这限制了从获得的各类Hi-C数据中准确识别染色质高级结构特征和潜在调控元件的效率。
2024年12月30日,国家生物信息中心徐晨欢研究团队在Nature Communications 在线发表题为“Footprint-C reveals transcription factor modes in local clusters and long-range chromatin interactions”的研究论文,该研究利用脱氧核糖核酸酶I(DNase I)消化基因组,开发了新一代Hi-C技术Footprint-C,在转录因子印迹(footprint)分辨率下,解析了转录因子局部或远程互作的新模式。
研究人员利用Footprint-C技术研究了K562和HEK293T两种细胞系的染色质构象,分别获得了19.3和9.3亿对染色质互作关系(chromatin contact)。在局部染色质水平,研究人员发现同一转录因子单独结合或与其它转录因子共结合的偏好在不同细胞系中是保守的,而多种转录因子共结合的方式却呈现细胞类型特异性。在染色质互作水平,Footprint-C相比目前最主流的Micro-C技术,在相同测序量下能鉴定出约1.5倍的染色质环(chromatin loop)和染色质条纹(chromatin stripe)结构。
研究人员基于Footprint-C解析出的转录因子印迹分辨率下的染色质相互作用,对不同转录因子间的互作进行了分析,发现CTCF-CTCF相互作用更多地介导远程互作,而MAZ-MAZ相互作用则更多地介导局部互作。研究人员进一步构建了一种新型的以特定转录因子motif为基本单位的全基因组染色质互作图谱。该图谱可以成功解析基因组上相距只有100 bp的两个CTCF motif分别与其它顺式元件发生的相互作用,这远远超越了常规Hi-C互作图谱在同等测序量下可以实现的分辨率。最后,利用这个携带转录因子“身份”信息的高清染色质互作图谱,研究人员还鉴定了涉及多重转录因子相互作用的多元染色质互作关系。
综上,Footprint-C构建的基于转录因子印迹的高分辨率染色质互作图谱,揭示了转录因子间的相互作用无论在基因组局部共结合水平,还是在染色质远程互作介导染色质高级结构方面,都具有丰富的调控模式,暗示了转录因子间的互作可能存在一套复杂的语法关系,用于实现对基因组复杂生命活动的调控。
国家生物信息中心徐晨欢研究员为本文的通讯作者,博士研究生刘晓昆和隗涵涵为本文的共同第一作者。该研究得到了国家自然科学基金、国家重点研发计划等项目的资助。
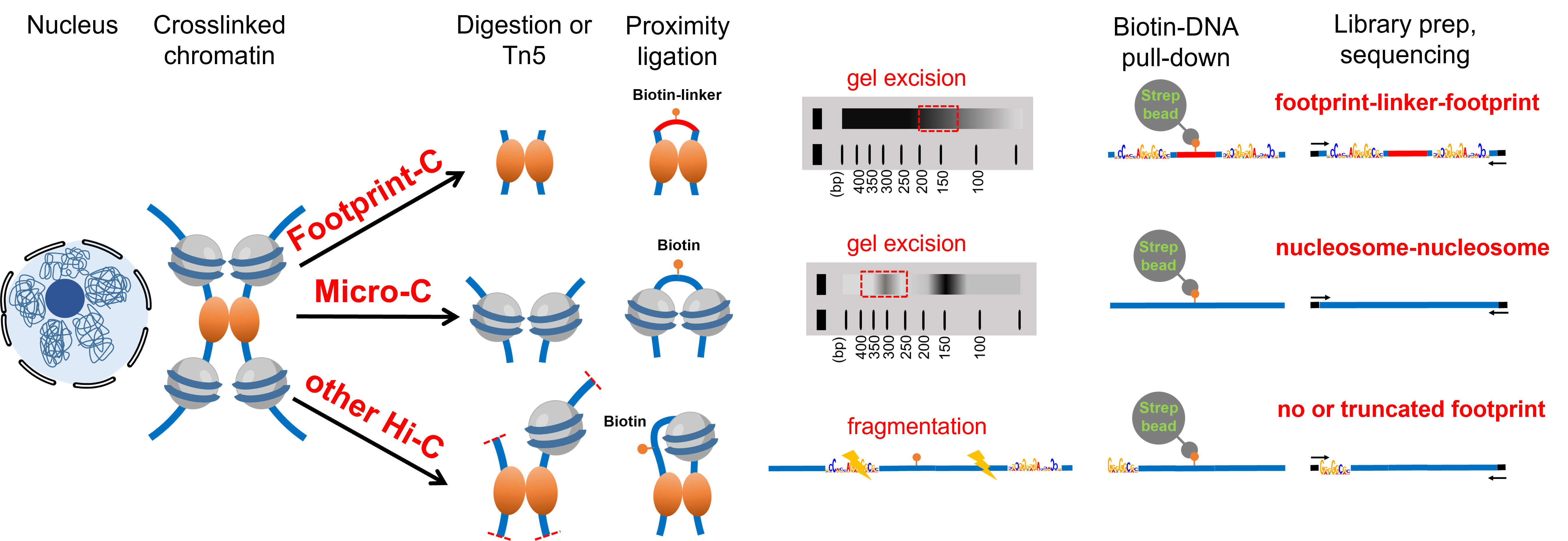
Footprint-C和其它主流Hi-C方法的原理比较






