国家基因组科学数据中心
一、中心成立及定位
国家基因组科学数据中心(National Genomics Data Center,简称NGDC)于2019年6月经科技部、财政部通知公布,由中国科学院北京基因组研究所(国家生物信息中心)作为依托单位,联合中国科学院生物物理研究所和中国科学院上海营养与健康研究所共同建设。中心面向我国人口健康和社会可持续发展的重大战略需求,建立生命与健康大数据汇交存储、安全管理、开放共享与整合挖掘研究体系,研发大数据前沿交叉与转化应用的新方法和新技术,建设支撑我国生命科学发展、国际领先的基因组科学数据中心。

中心定位与目标
二、中心运行机制与组成
中心主任:赵文明正高级工程师
中心副主任:章张研究员、宋述慧研究员
工作团队:组学原始数据归档库、基因组数据库、基因组变异数据库、基因表达数据库、表观基因组数据库、非编码RNA数据库、精准医学知识库、生物信息工具库和系统运维部等。

中心集体照
三、年度主要科研进展
1. 多组学数据资源体系持续拓展和更新
国家基因组科学数据中心持续拓展和更新多组学数据资源体系。2023年重点加强多组学数据整合、知识融合、新库开发,以及核心数据库升级。其中,新开发了多个数据库,包括原生生物(P10K)、细菌(NTM-DB, MPA)、植物(PPGR, SoyOmics, PlantPan)和疾病/性状关联(CROST, HervD Atlas, HALL, MACdb, BioKA, RePoS, PGG.SV, NAFLDkb)等数据资源。截至2023年12月底,已支持各类科技项目17,000多个,汇交数据量达40PB,相关数据已在572种国内外期刊的3,000多篇文章发表,为国家基因组科学数据的汇交共享、安全管理和挖掘利用提供了重要支撑。数据库建设整体情况以“Database resources of the National Genomics Data Center, China National Center for Bioinformation in 2024”为题在Nucleic Acids Research 在线发表。
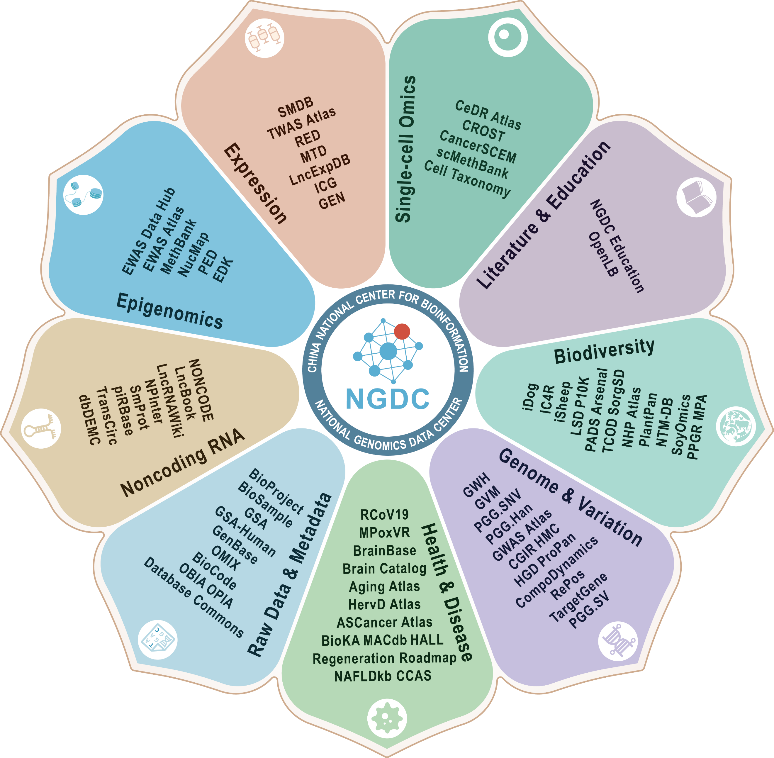
国家基因组科学数据中心多组学数据资源体系
2. GSA数据库入选全球核心生物数据资源
2023年12月11日,国家基因组科学数据中心建设的组学原始数据归档库(Genome Sequence Archive, GSA)成功入选由国际生物数据联盟(Global Biodata Coalition, GBC)发起的全球核心生物数据资源(Global Core Biodata Resource, GCBR)。GCBR现收录52个国际数据库,GSA是我国目前唯一入选的数据库。作为生命组学原始测序数据汇交、存储、管理和共享的公益性数据库,GSA旨在推动全球生命组学数据的共享与应用。此次入选GCBR有利于促进我国生命科学组学数据的统一管理与开放共享,推动与国际社会的深度交流合作,并加速我国在大数据时代的生命科学研究进程。

GSA入选GCBR
3. 全球生物数据库目录Database Commons入选2022年度“中国生物信息学十大进展”
生物数据库作为全球各类生命科学研究的基础支撑,极大促进了大数据向知识的转化,并推动了众多研究领域的重要创新。NGDC自2015年起建设全球生物数据库目录Database Commons,联合国内外多家科研机构,持续开展数据积累和功能完善。截至2023年底,已审编收录76个国家/地区2,142家机构发布的6,380个数据库。同时,创新设计了z-index用于评估数据库的科学影响,并根据数据库文章引用和z-index对生物数据库及其隶属机构和国家进行排名。Database Commons提供了全球生物数据库的系列统计数据和趋势,为更好地了解数据库发展态势及其对生命健康科学的影响提供全球视角。该成果以“Database Commons: a catalog of worldwide biological databases”为题在 Genomics Proteomics Bioinformatics 在线发表,并入选2022年度“中国生物信息学十大进展”。

Database Commons入选2022年度“中国生物信息学十大进展”
4.发布基因序列数据库GenBase
基因的序列和注释信息(包括DNA、RNA和蛋白序列信息)是支撑基因功能研究的核心基础数据之一。为保障我国基因序列数据的主权和安全,满足我国科研人员在基因序列数据汇交、管理和共享过程中的现实需求,NGDC开发了基因序列数据库GenBase,于2023年3月正式上线,为用户提供基因序列数据汇交共享和查询下载服务。GenBase对标美国国家生物信息中心NCBI的GenBank数据库,立足中国,服务全球,可接收来自全球科研人员的数据提交,并且通过数据交换机制实现与GenBank的无缝共享。
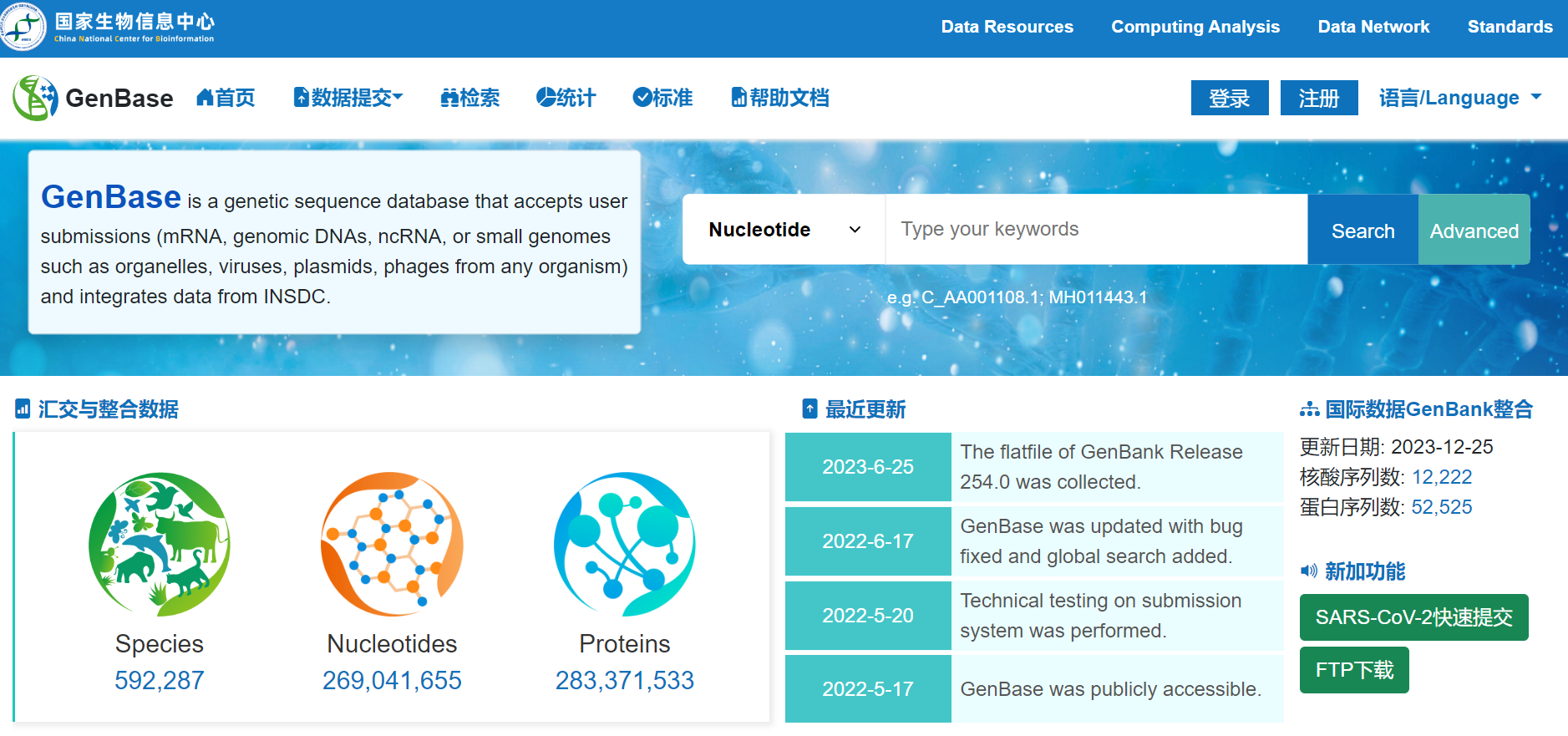
GenBase网站页面
5.2019新冠病毒信息库(RCoV19)持续升级更新
2023年RCoV19进一步升级,开发了全自动化的数据智能审编模型和数据共享页面,建立了基因组快速变异解析流程、单倍型网络演化构建算法以及基于机器学习的高风险株系预警模型,开发了新冠病毒传播演化实时监测平台、高风险变异株预警可视化系统和交互式突变谱快速比对功能模块,实现了新冠病毒基因组序列、变异和演化支系的可视化动态监测,高风险变异株的及早预警,以及重要序列或谱系的变异特征规律分析,成为集新冠病毒基因组数据自动整合、变异监测、风险预警和突变效应知识于一体的全链条综合性平台。截至2023年12月25日,RCoV19已收录新冠病毒序列超1,700万条,为全球182个国家/地区400多万名访客提供数据服务,累计数据下载达190多亿条。该成果以“RCoV19: a one-stop hub for SARS-CoV-2 genome data integration, variant monitoring, and risk pre-warning”为题在Genomics Proteomics Bioinformatics 在线发表。
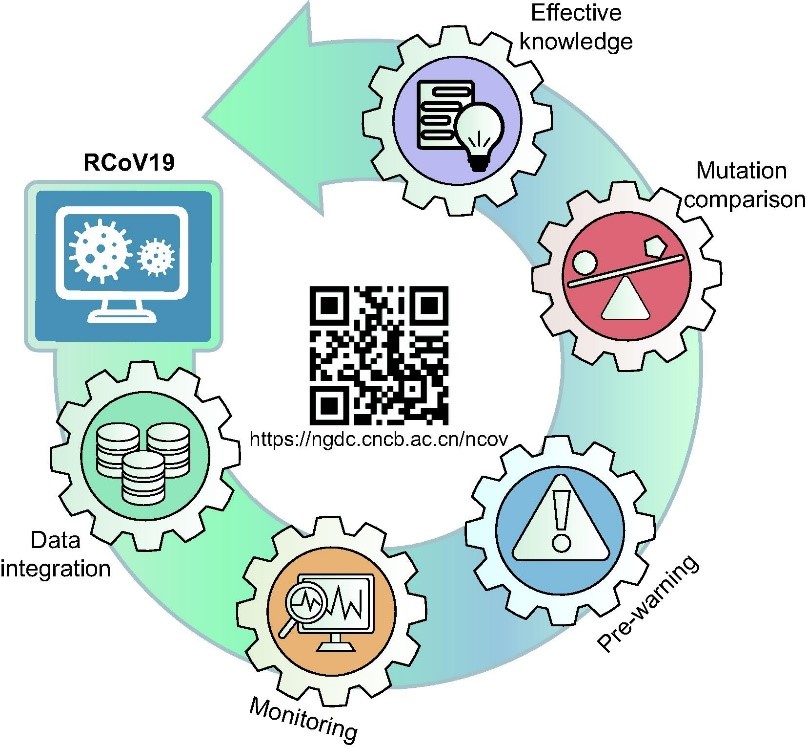
RCoV19一站式平台
6.开发人类癌症代谢物关联知识库MACdb
随着代谢组学研究的发展,针对不同癌症类型、基因组异常、药物反应评估的代谢物关联关系已被广泛报道。MACdb是一个基于人工审编的知识库,用于收录代谢产物与癌症之间的关联关系。目前已整合基于269个癌症特征的40,710个关联关系,涵盖17类高发病率或高死亡率的癌症,是当前涵盖癌症类型最全的癌症—代谢物关联知识库。MACdb提供直观的浏览功能及多维度关联检索,通过知识图谱实现对癌症、特征和代谢产物间整体情况的展示。此外,NameToCid和Enrichment工具可用于标准化代谢物及富集代谢产物与各种癌症类型和特征的关联。该成果以“MACdb: a curated knowledgebase for metabolic associations across human cancers”为题于2023年7月在Molecular Cancer Research正式发表,并被选为该刊当期封面故事。

MACdb知识库入选MCR期刊封面故事
7.开发人类内源性逆转录病毒相关疾病知识库HervD Atlas
人内源性逆转录病毒(HERVs)是远古时期外源性逆转录病毒感染宿主生殖细胞或胚胎干细胞并整合到人类基因组上的前病毒序列,近年研究表明其在正常生理和病理发展等重要生命过程中发挥重要作用。为此,NGDC与本所陈非团队合作开发了人类内源性逆转录病毒相关疾病知识库HervD Atlas,整合250多篇HERVs相关疾病研究文献数据,通过人工审编获得60,726条高质量的HERVs与疾病关联条目,涵盖21,790种HERVs,149种疾病和610个受影响基因。该数据库系统整合HERVs、疾病和基因的关联信息,构建了交互式知识图谱,为关联知识整合及推断提供了界面友好的可视化平台。该成果以“HervD Atlas: a curated knowledgebase of associations between human endogenous retroviruses and diseases”为题在Nucleic Acids Research 在线发表。
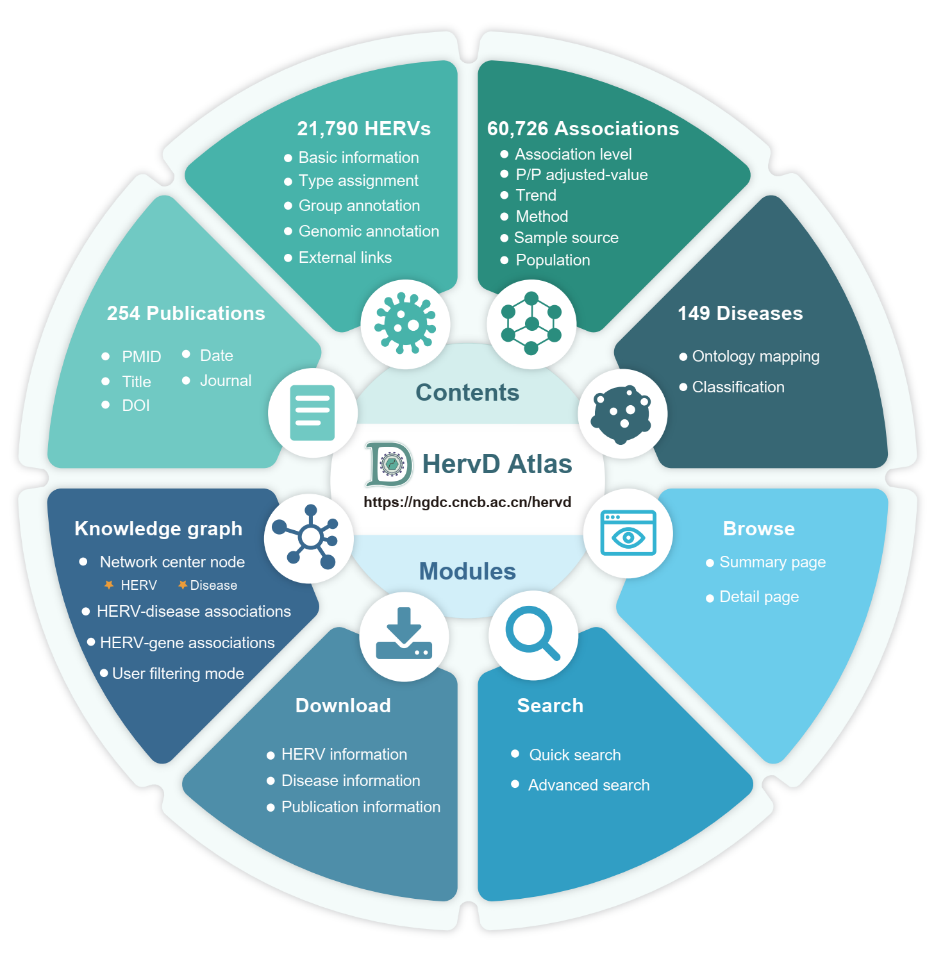
HervD Atlas概览
8.发布生物标志物知识库BioKA
生物标志物(Biomarker)不仅是诊断分析发展、确定新药研发靶标的基础,也是培育新品种的基础,在个性化医疗、药物研发、临床护理和分子育种等多个领域发挥重要作用。为此,NGDC开发了生物标志物知识库BioKA,从4,747篇文献中人工审编与整合了人和30个动物物种总共951个疾病/性状相关的16,296个生物标志物,并提供了经过标准化后的308个品种以及相应的生物标志物信息。BioKA不仅丰富了人类标志物信息,也填补了已有的生物标志物数据资源在动物疾病和动物分子育种方面的空缺。该成果以“BioKA: a curated and integrated biomarker knowledgebase for animals”为题在Nucleic Acids Research 在线发表。
9.开发空间转录组综合资源存储库CROST
随着空间转录组测序技术的发展,空间转录组数据的激增急需一个用户友好的数据库系统,以便于轻松访问数据,并进行可视化和个性化分析。为此,NGDC与本所方向东团队合作开发了空间转录组综合资源存储库CROST,应用标准化处理流程整合了182个高质量的空间转录组数据集,涵盖8个不同物种、35种组织类型和56种疾病的1,033个子数据集。针对单个样本提供了全面的生物信息分析,包括空间变异基因(SVG)分析、细胞类型注释、空间相关性、空间共定位、通讯分析和功能注释等。CROST通过集成空间转录组、经典转录组、表观基因组和基因组的数据全面阐明了肿瘤相关SVG,是用户(尤其是临床医生)快速评估特定癌症类型中基因表达水平、甲基化水平、拷贝数变异以及预后的宝贵工具。该成果以“CROST: a comprehensive repository of spatial transcriptomics”为题在Nucleic Acids Research在线发表。
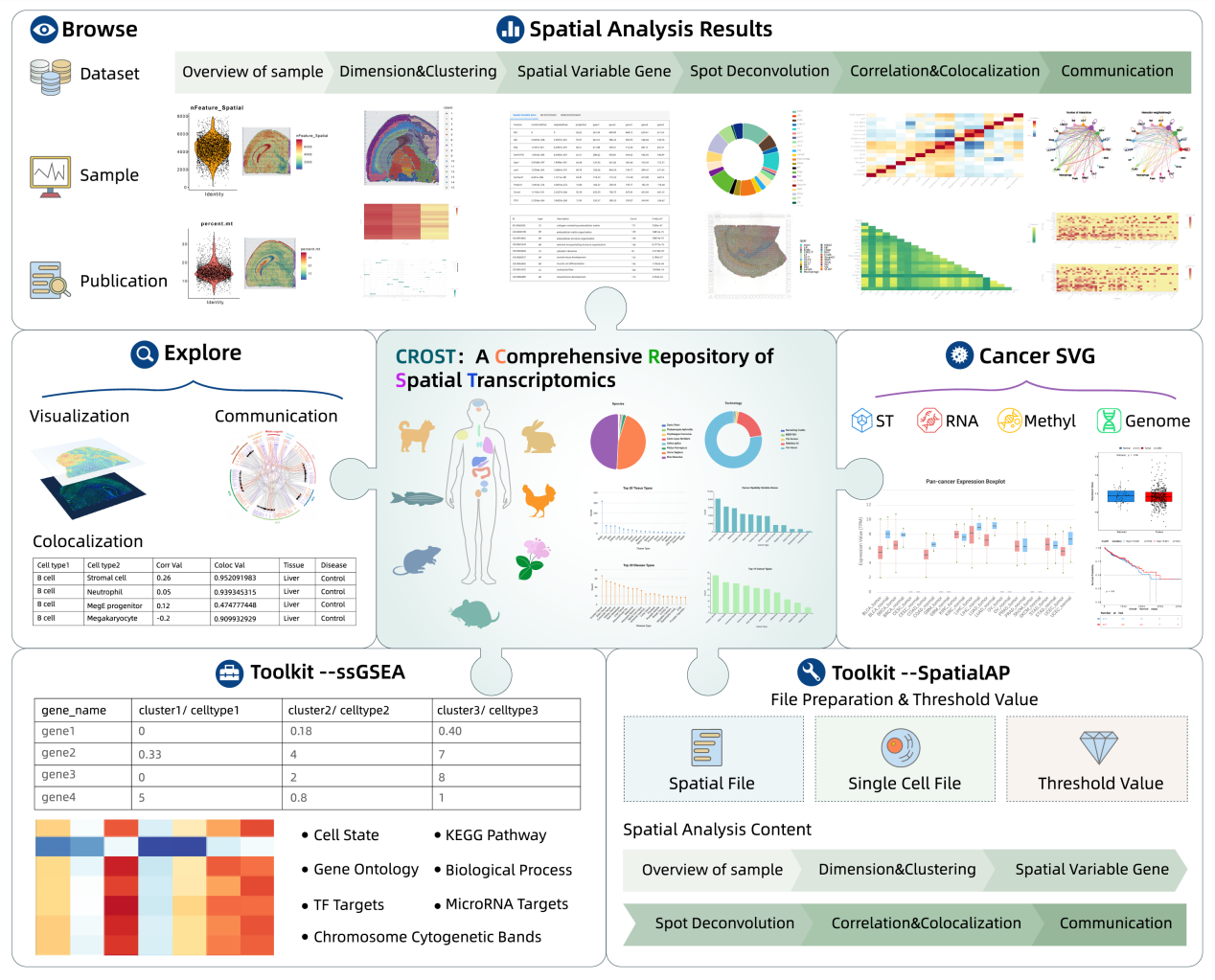
CROST概览
10.发布开放生物医学影像存档库OBIA
生物医学影像数据中包含大量的隐私信息,如何构建生物医学影像数据管理平台,既保障数据隐私信息的安全,又能促进全球数据的共享,是当前生物医学影像数据使用中急需解决的问题。为此,NGDC与中国人民解放军总医院第七医学中心合作开发了开放生物医学影像存档库OBIA,向国内外科研人员提供医学影像数据递交、归档、发布与共享的公共服务。为保障影像数据中隐私信息的安全,OBIA制定了统一的去识别和质量控制流程,并设置了开放访问和受控访问两种不同类型的数据访问策略。目前OBIA收录的影像数据包含子宫内膜癌、卵巢癌和宫颈癌三大妇科肿瘤,来自4,136项研究的937个个体,包括24,701个系列和1,938,309幅影像,涵盖了9种模态和30个解剖部位。该成果以“OBIA: an open biomedical imaging archive”为题在 Genomics Proteomics Bioinformatics 在线发表。
11.开发大豆多维组学数据库SoyOmics
高通量测序技术的发展促使大豆组学研究不断深入。实现大豆多维组学数据的整合分析,将为大豆遗传育种提供有力支持。为此,NGDC与中国科学院遗传发育所田志喜团队合作开发了大豆多维组学数据库SoyOmics。该库目前收录了27个大豆品系的从头组装基因组数据,并对相应基因组信息进行了全面的基因组注释,从基因组、变异组、转录组、表型组等不同层面整合了大豆相关数据集,实现了不同层次组学数据的交互查询和联合比较分析,为大豆遗传学及育种研究提供基础数据支撑和全新的观察视角。该成果以“SoyOmics: a deeply integrated database on soybean multi-omics”为题在Molecular Plant 在线发表。
12.发布热带作物组学数据库TCOD
测序技术的飞速发展推动了热带作物研究领域里程碑式的发展,积累了海量的多组学数据,然而,大量的数据分散在不同的数据中心或网站,给数据利用带来了不便,亟需开发一个综合数据整合与共享平台。为此,NGDC与海南大学王文泉团队等合作开发了热带作物组学数据库TCOD(Tropical Crop Omics Database)。目前TCOD已整合15种热带作物的基因组、变异组、转录组和品种数据,以基因为桥梁关联多种组学数据,为用户提供便捷的数据浏览、检索和下载等服务。TCOD不仅提供了物种间的同源基因关系用于跨物种功能探索,还提供了一系列在线工具用于数据挖掘,为热带作物选择育种和性状改良研究提供支撑。该成果以“TCOD: an integrated resource for tropical crops”为题于2023年10月在Nucleic Acids Research 在线发表。
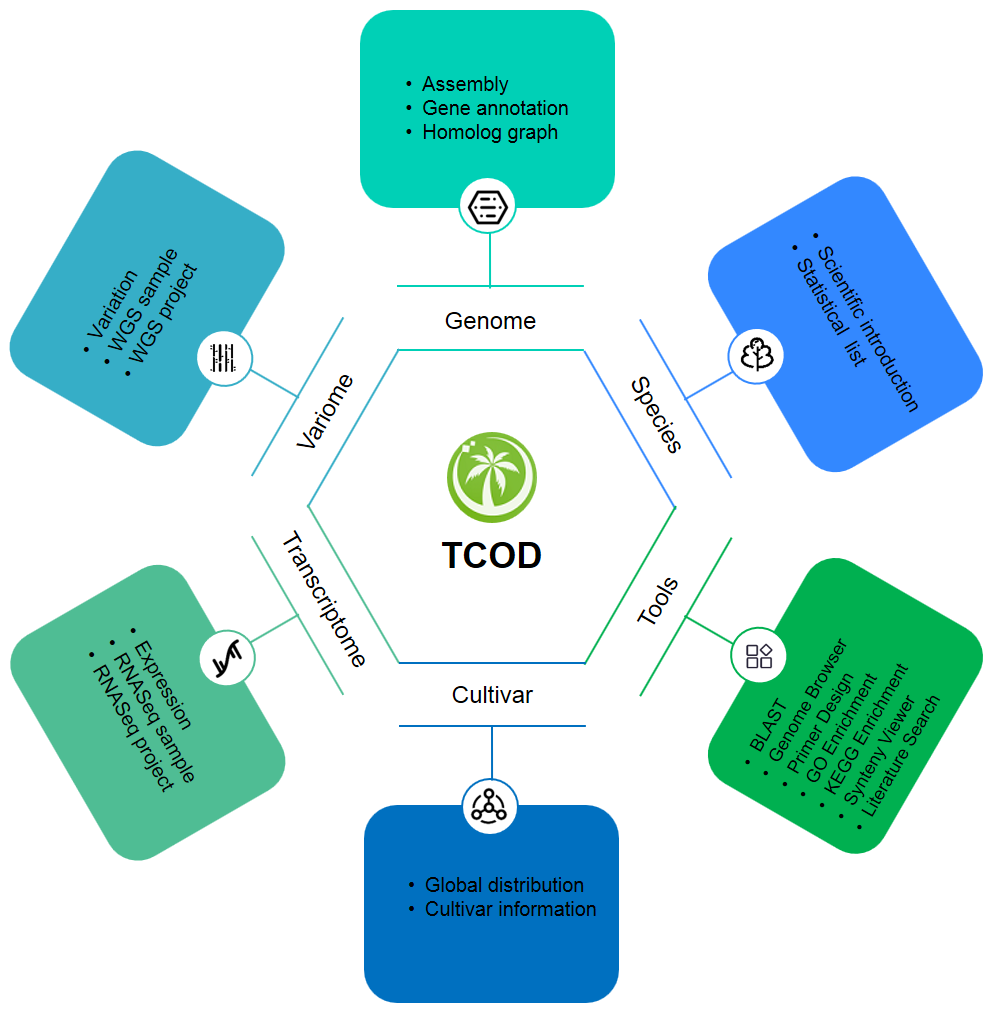
TCOD数据库概览
13.开发多年生木本植物基因组与调控信息库PPGR
多年生木本植物是林业作物中重要的植物类群,其生命周期长,基因组大且杂合度高,具有独特的生理代谢途径和胁迫抵抗特性。全面整合多年生木本植物组学数据资源,建立系统的遗传调控网络,对于阐明该植物类群的关键生物学过程和独特性状具有重要意义。为此,NGDC与北京林业大学谢剑波团队合作开发了多年生木本植物基因组与调控信息库PPGR。该信息库是首个专注于多年生木本植物的在线资源平台,目前已整合60种重要多年生木本植物的基因组数据,应用标准化流程分析了9,016个植物转录组样本,鉴定了107,344个转录因子、10,263个抗病基因以及53,829个水平转移基因,系统构建了多维基因调控网络,将为林木基因组学和基因调控研究领域科研突破和发现提供强大的数据支持和信息保障。该成果以“PPGR: a comprehensive perennial plant genomes and regulation database”为题在Nucleic Acids Research在线发表。
14.开发植物图像及相关性状开放归档库OPIA
随着高通量植物表型采集技术在植物表型组学研究中的广泛应用,产生了大量的图像和基于图像的性状数据,这些数据是种质筛选、植物病虫害鉴定、农艺性状挖掘等应用的重要资源。为此,NGDC与中国科学院遗传发育所胡伟娟团队合作开发了植物图像及相关性状开放归档库OPIA,为国内外科研人员提供植物图像及相关性状数据递交与共享的公共服务。OPIA采用标准化人工审编流程整合了56个高质量的植物图像数据集,涵盖11个物种、6种组织类型,总计566,225张图像、2,417,186个注释实例。通过对来自不同传感器类型的图像样本及相应标签数据的运用,有利于促进研究人员进一步提高智能预测方法的精度,揭示植物生长的动态规律,进而推动全球植物表型组学领域的创新和发展。该成果以“OPIA: an open archive of plant images and related phenotypic traits”为题在Nucleic Acids Research在线发表。
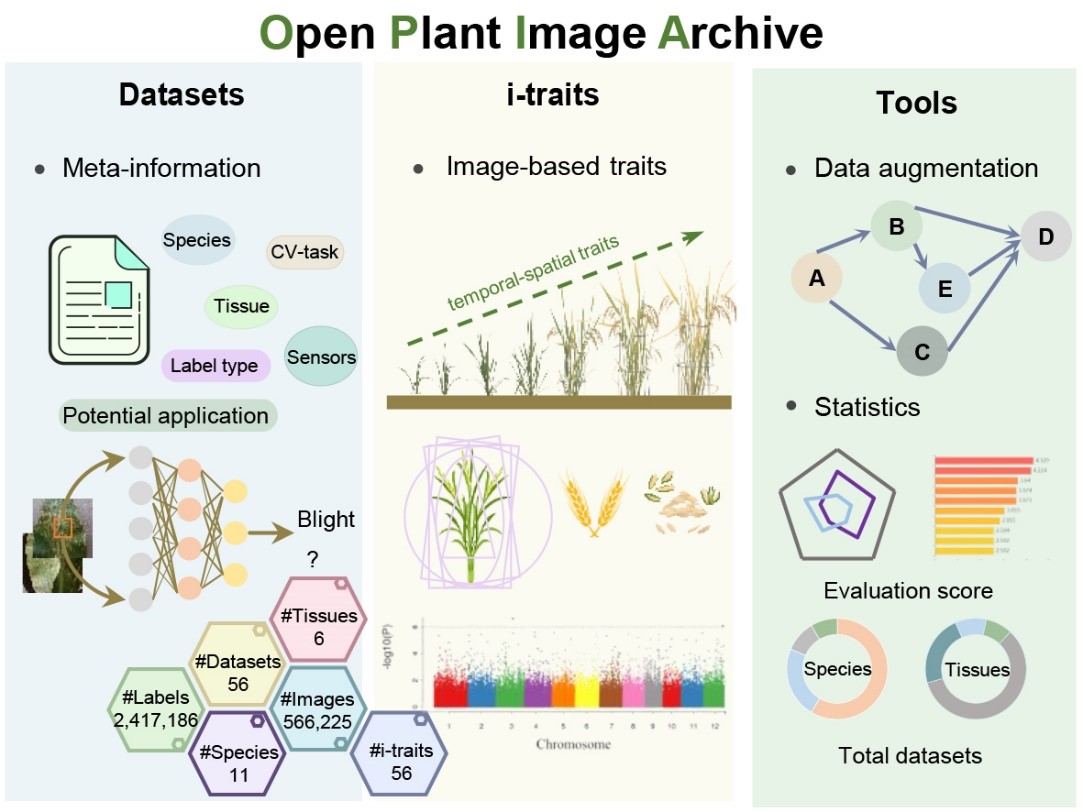
OPIA功能概览
四、获奖与荣誉
国家基因组科学数据中心荣获2023年北京市朝阳区“最美科技创新团队”
全球生物数据库目录Database Commons入选2022年度“中国生物信息学十大进展”
鲍一明研究员荣获“全国归侨侨眷先进个人”
赵文明正高级工程师荣获“中国科学院优秀党务工作者”
马利娜副研究员荣获2023年度中国科学院青促会优秀会员
陈梅丽高级工程师入选2023年度中国科学院技术支撑人才
2023年度研究生国家奖学金:宗文婷、麦嘉琳
2023年度中国科学院朱李月华优秀博士生奖:李昭






