国家生物信息中心发布更新版癌症单细胞表达图谱数据库CancerSCEM 2.0
近日,国家生物信息中心开发的癌症单细胞表达图谱数据库CancerSCEM 2.0正式上线。该研究内容以“CancerSCEM 2.0: an updated data resource of single-cell expression map across various human cancers”为题在国际学术期刊Nucleic Acids Research 在线发表。
近年来,单细胞转录组测序(scRNA-seq)技术不断革新,相应的生物信息学分析工具也呈现蓬勃发展态势,有力支撑了研究人员将scRNA-seq数据转化成生物学或医学相关信息。scRNA-seq技术已广泛应用于肿瘤学研究,通过鉴定不同细胞亚群、剖析肿瘤微环境以及表征单细胞基因表达变化,帮助研究人员深入理解复杂的肿瘤生物学。此外,癌症的代谢重编程研究近几年逐渐兴起,作为癌症的核心生物学标志之一,已有许多代谢靶向药物进入临床试验阶段。因此,对海量癌症scRNA-seq数据进行标准化整合分析对于更好地支撑癌症相关研究以及临床应用至关重要。自2022年首次发布以来,CancerSCEM数据库在癌症样本的单细胞基因表达定量、细胞类型精确注释以及免疫反应研究等方面得到了广泛应用。
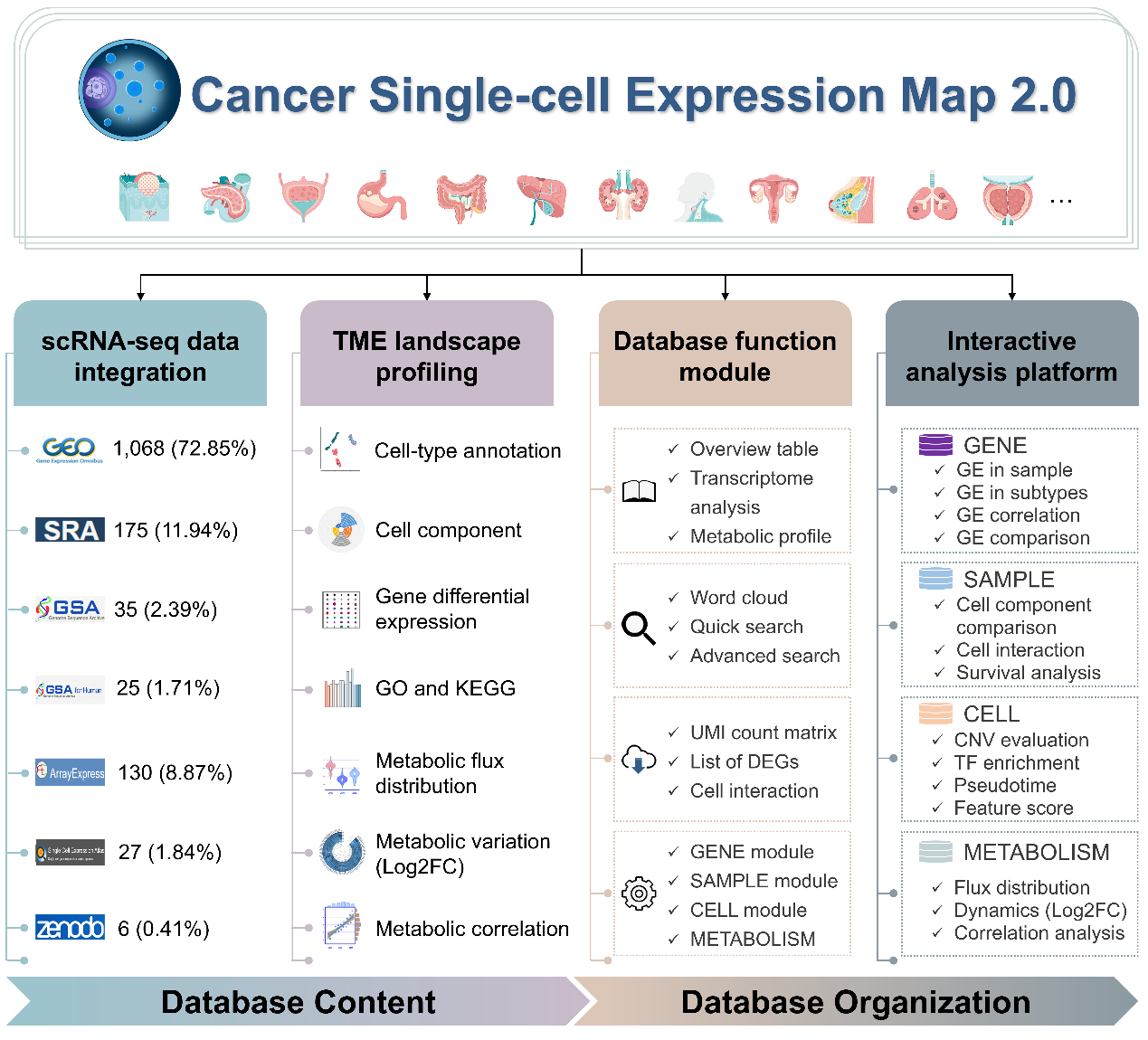
CancerSCEM 2.0版本于2024年6月公开上线,共收录1,466套癌症相关scRNA-seq数据集及其标准化分析结果,涵盖127个研究项目、74种人类癌症类型以及8种单细胞测序方法,并首次纳入正常组织样本与健康的外周血样本,可用作正常对照支撑用户开展下游比较分析。CancerSCEM 2.0从多个层面进一步增强了数据分析及可视化功能,首先在转录组层面新增了基因组拷贝数变异评估(inferCNV)、转录因子富集(pySCENIC)、拟时序构建(monocle2)以及7项重要生物学特征打分(毒性、炎症、压力等),所有分析结果均可通过交互式分析平台获取。同时,开展了以样本为单位的168个代谢模块和34个KEGG代谢通路的单细胞代谢通量推断、跨细胞类型代谢动态变化追踪以及代谢相关性评估等分析内容,并设置全新的代谢浏览页面为用户提供系列可视化分析结果。此外,与1.0版本相比,CancerSCEM 2.0整合了来自33个TCGA癌症研究项目涵盖11,167名患者的基因表达数据,进一步提升了数据库在细胞群体水平的基因表达概览以及在线生存分析功能。
CancerSCEM2.0数据库提供以scRNA-seq数据集为核心单元的网站系统,配备用户友好的数据浏览、检索、可视化及下载功能。除1.0版本的GENE与SAMPLE模块以外,CancerSCEM 2.0对交互式在线分析平台进行了系统升级与扩展,从头研发和部署了两个全新的分析模块——CELL与METABOLISM,二者将分别提供新增的4项转录组分析与3项代谢相关分析功能,均可实现秒级别的实时分析及可视化。用户可借助此平台开展多维度定制分析,深入探索新的潜在有效的癌症类型特异或细胞类型特异的生物标志物。
国家生物信息中心高级工程师曾瀞瑶、博士研究生聂致、尚云飞、麦嘉琳为本文共同第一作者,肖景发研究员为通讯作者。该研究得到了中国科学院青年创新促进会、中国科学院战略性先导科技专项以及国家自然科学基金的资助。
CancerSCEM 2.0 数据标准化处理与数据库功能模块概览






