国家生物信息中心徐晨欢研究组揭示转录因子在天然染色质上的动态量化特征
转录因子在染色质上的单次驻留时间通常为几秒至几十秒。驻留时间的动态变化会影响基因转录或染色质高级结构。使用甲醛交联来固定细胞核中蛋白质-DNA相互作用的染色质免疫共沉淀(ChIP)技术是检测转录因子在染色质上驻留水平的常用方法。然而,通常持续10分钟的甲醛交联会以累积的方式固定短时的转录因子-DNA相互作用,从而扭曲转录因子在不同结合位点驻留时间的差异,扰乱对转录因子真实驻留水平的测定。在未经任何化学交联的天然染色质(native chromatin)上测定转录因子的驻留能力,有助于解析更量化的转录因子动态驻留特征及其生物学意义。
5月14日,国家生物信息中心徐晨欢研究组在Molecular Systems Biology 期刊在线发表了题为“A continuum of zinc finger transcription factor retention on native chromatin underlies dynamic genome organization”的研究论文。该研究基于优化的微球菌核酸酶(MNase)消化,开发了loMNase-seq和N-ChIP技术,以高分辨率转录因子足迹(footprint)的形式,分别测定了天然染色质上全局或特定转录因子(CTCF、MAZ、ZNF143等)的驻留位点与驻留水平。
单次ChIP实验只能提供特定实验条件下染色质上转录因子驻留的信息。利用天然染色质可以实时响应外部环境、动态变化的特性,研究人员在连续增强的NaCl浓度下开展了一组N-ChIP实验,发现连续增强的盐浓度可以实现从天然染色质上连续“剥离”转录因子CTCF或MAZ的结合,表现为更高盐浓度的N-ChIP捕获的转录因子驻留位点是更低盐浓度N-ChIP捕获的转录因子驻留位点的子集。利用在这个盐浓度梯度下测定的天然染色质上CTCF的结合信号,研究人员进一步建立了不同CTCF基序(motif)的“耐盐得分”——S-score,作为反映CTCF蛋白在天然染色质不同基序上实时驻留能力的量化指标。与从交联ChIP,以及其它天然染色质相关实验(CUT&RUN,CUT&Tag等)获得的CTCF量化驻留信息相比,S-score可以更好地解释这些CTCF基序的序列特征、CTCF驻留对锌离子剥夺等应激条件的耐受能力,以及由这些CTCF基序介导的染色质环(chromatin loops)等高级结构在锌离子剥夺条件下的差异稳定性。这充分展现了使用N-ChIP技术在天然染色质上研究染色质高级结构实时变化规律的巨大优势。
综上,该研究定量测定了CTCF、MAZ等转录因子在天然染色质上的驻留水平,揭示了天然染色质上转录因子的差异驻留水平是染色质高级结构动态特征的基础,为转录因子调控领域的研究提供了两个新的实验工具,以及一套以递增盐浓度为基础的定量分析方法。
国家生物信息中心徐晨欢研究员为本文的通讯作者,博士研究生胡思玲和刘扬影为本文的共同第一作者。该研究得到了国家自然科学基金、国家重点研发计划等项目的资助。
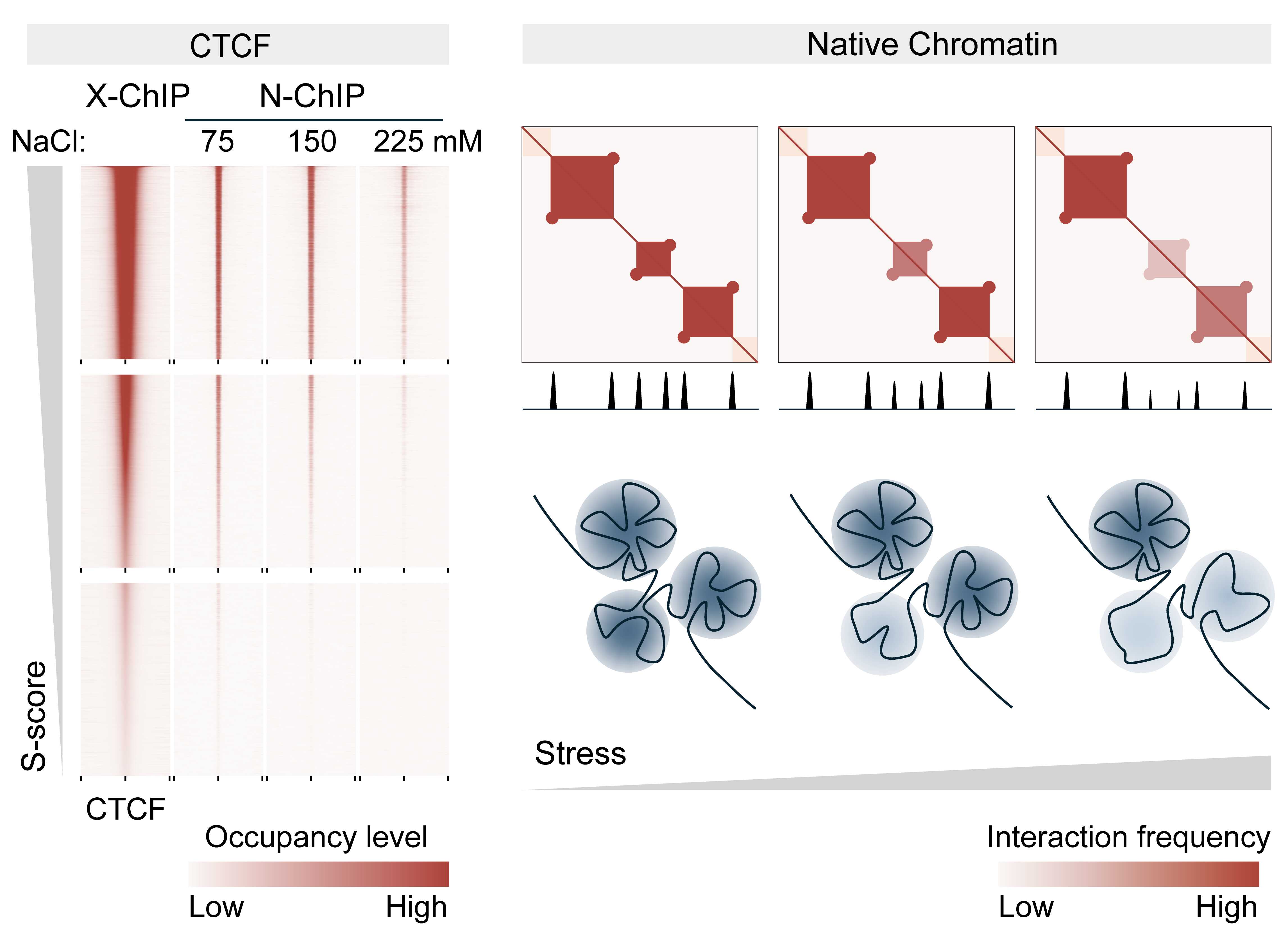
天然染色质上转录因子的差异驻留水平构成染色质高级结构动态特征的基础






