国家生物信息中心开发通用型高通量单细胞多组学新技术
细胞是生命的基本单元。近年来,单细胞组学技术的快速发展前所未有地革新了人类对细胞多样性和异质性的认知,推进了生物医学重大规律的发现,为建立可预测可编程的虚拟细胞基础大模型提供语料。细胞图谱技术被Nature 期刊列为2024七大关注技术之一。2024年11月,国际人类细胞图谱计划在Nature 期刊及其子刊发表了40余篇论文合集,标志着“细胞身份普查”取得了阶段性成果。下一阶段的重心,则是单细胞多模态和人群尺度的研究,深入解析遗传背景、环境、病理条件与性状和表型之间的复杂互作。然而,当前的单细胞多组学技术的成熟度严重滞后于单细胞转录组技术,在人群队列的大规模研究中面临着通量、普适性等诸多挑战,亟待突破。
近日,国家生物信息中心计算生物学部蒋岚团队在Nature Methods 期刊发表了题为 UDA-seq: universal droplet microfluidics-based combinatorial indexing for massive-scale multimodal single-cell sequencing的研究论文。该工作报道了自主研发的新型单细胞多组学测序技术UDA-seq,基于组合标记技术路线优化升级广泛使用的液滴微流控平台,成功实现细胞通量和假单细胞率的“解偶联”,从而突破了通量限制。该技术是广谱型的通量提高策略,支持常见的单细胞多组学,例如同一细胞的RNA和VDJ共检测、RNA和ATAC共检测,以及RNA和CRISPR扰动共检测等。在兼容多模态的同时将单通道细胞通量提高到刷新记录的10万细胞以上,比现有技术提升了10-20倍。同时,利用遗传多样性结合生物信息学拆解,以免标记的方式实现了单通道20-40例人类样本的多路复用,大幅降低单例样本的数据成本。此外,还形成了大规模的从微量冰冻血液样本和微量冰冻活检样本中获取高质量、多模态、单细胞精度数据的完整解决方案。
研究团队将UDA-seq 应用于三个应用场景中,包括衰老、疾病人群队列研究以及高通量CRISPR筛选研究。首先,对健康人和多种疑难肾病患者的肾脏组织构建了单细胞RNA和ATAC双组学图谱。克服了微量冰冻样品前处理的难点,每例样本仅需五分之一条针穿活检样品,在液滴微流控中利用2个通道的反应收集到来自35例捐献者的一共20万高质量双模态单细胞数据。通过建立新算法,鉴定出与蛋白尿和肾损伤表型相关的罕见细胞类型POD细胞和EC-GC细胞,以及关键细胞亚群的调控网络和关键因子。利用UDA-seq,研究者还构建了自然衰老人群队列的PBMC单细胞转录组和免疫受体序列双组学图谱。利用2个通道的反应,收集到12万高质量单细胞数据,解析了衰老相关的细胞亚型,并报道了衰老相关的新细胞亚型:ITGB1+PREX1+ Naive CD4+ T细胞。最后,UDA-Seq也展示了对高通量CRISPR筛选的兼容,在胃癌细胞系中系统解析了细胞对溴结构域蛋白基因家族扰动的响应。
综上,该工作突破现有技术的瓶颈,在兼容多模态的同时大幅提高了单细胞测序的通量,在真实世界样本中证明了可靠性和高质量,实现了低成本的高效数据生产。开发了新算法鉴定临床表型相关的关键细胞亚群、调控网络和关键因子,为人群尺度健康队列和疾病(例如癌症,自身免疫,感染等)队列细胞图谱大规模研究建立了一个新型研究范式。此外,该工作集成常见可及的液滴微流控平台改造提升,有望大规模推广,填补多模态、扰动前后单细胞数据集的匮乏,为下一代的多模态虚拟细胞基础大模型提供数据支撑。
上述工作由国家生物信息中心蒋岚团队和多家单位合作完成。蒋岚研究员、北京协和医院陈丽萌教授、中国科学院动物研究所刘光慧研究员、国家生物信息中心张维绮研究员、衢州市人民医院张峰教授、北京大学舒绍坤教授为本文的共同通讯作者。蒋岚团队特别研究助理李芸、博士研究生黄正,北京协和医院徐鲁斌医生,国家生物信息中心助理研究员范艳玲,衢州市人民医院助理研究员平骏为文章的并列第一作者。该研究得到国家重点研发计划、中国科学院先导专项、中国科学院全球共性挑战专项、国家自然科学基金委等项目的资助。
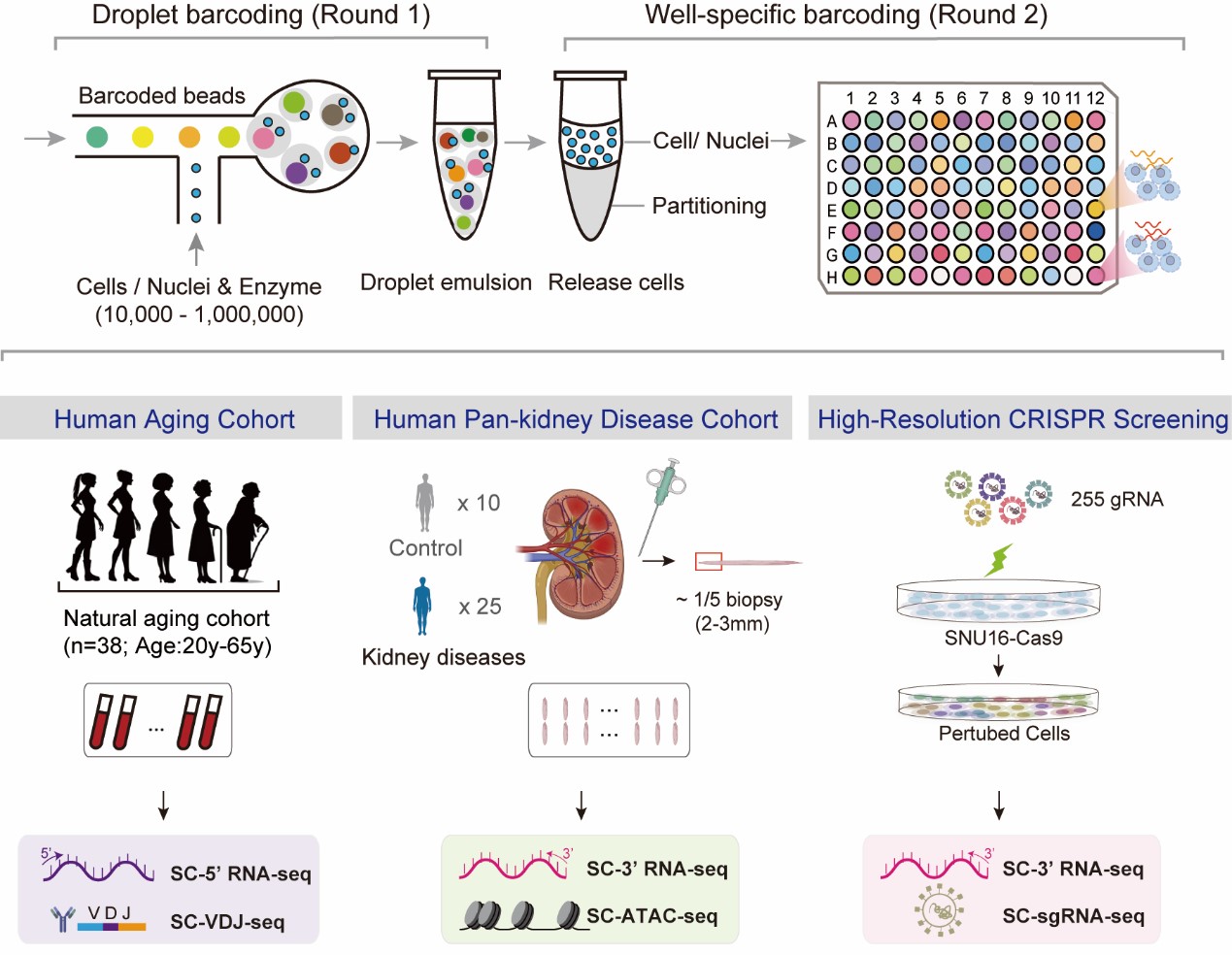
通用型高通量单细胞多组学技术UDA-seq 流程图及应用场景






