北京基因组所(国家生物信息中心)合作开发新冠病毒基因组浏览器
1月20日,中国科学院北京基因组研究所(国家生物信息中心)与中国科学院上海营养与健康研究所合作开发的新冠病毒基因组浏览器成果,以“Coronavirus GenBrowser for monitoring the transmission and evolution of SARS-CoV-2”为题在国际学术期刊Briefings in Bioinformatics 在线发表。
病毒演化关系及传播链条推演在疫情防控与病毒溯源中具有重要意义,随着新冠疫情的不断发展及新冠病毒基因组序列数量的快速增长,基于百万级病毒基因组数据的演化分析与展示变得极其困难。为此,中科院北京基因组所(国家生物信息中心)国家基因组科学数据中心赵文明团队和营养与健康所李海鹏、张国庆团队合作开发了Coronavirus GenBrowser(CGB)。基于分布式基因组序列比对,CGB采用自主开发的超快构树新方法进行进化树构建,并基于超快速极大似然法估算内节点时间,解决了快速构建百万病毒基因组序列的进化关系与可视化展示的关键问题。在撰写论文之前,CGB已于2020年8月正式上线并持续提供在线服务。
CGB是一个开放访问和下载的新冠基因组演化可视化工具,采取质控后的高质量新冠基因组序列数据以及传播相关的元数据构造了百万级别的进化树。同时,基于CGB特有的带有突变和日期注释的树结构,新产生的新冠病毒基因组序列可以超快速并准确地添加到进化树上形成新的进化树。CGB还提供基于树结构的基因组变异可视化展示。CGB用户界面友好,用户可以选择节点并高效地对进化树进行细节可视化和进一步分析,并且无需任何编程。CGB同时也提供了下游分析功能,包括变异分析、谱系追踪、检测分支特异的加速进化以及病毒变异正选择等功能。目前CGB核心数据文件仅24.6Mb,却包含了2.79百万新冠病毒全基因组变异及其元数据。通过团队持续排查元数据中可能包含的错误,CGB的数据接口提供了非常可靠的数据源。通过CGB双命名法,其1.15百万内枝均得以准确命名,并实现可搜索和查询。
为了便于用户使用和操作,CGB提供web版本,支持九种语言,同时提供war包供下载和在任何网站中迁移使用。为方便使用CGB的所有功能,推荐使用单机版(http://www.egps-software.net/egpscloud/eGPS_Desktop.html)。开发团队将进一步开发CGB 2.0,致力提供千万、乃至亿基因组级别的新冠病毒进化和传播分析,为国内、全球的防疫防控提供有力支持。
这一工作得到国家重点研发计划和中科院战略性先导科技专项的支持。
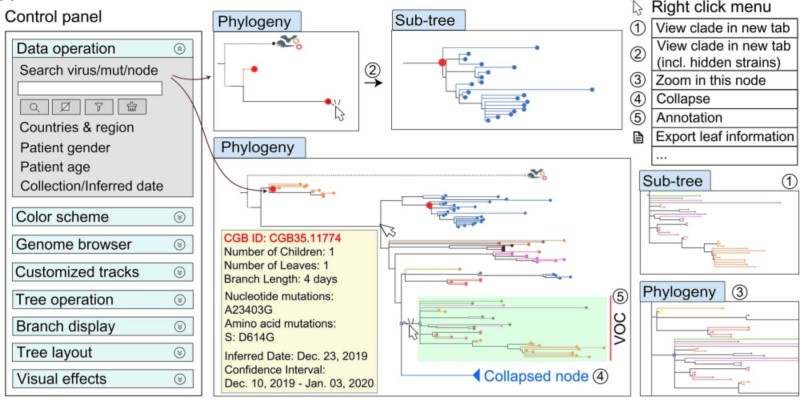
新冠病毒基因组浏览器的用户界面和操作示意






