北京基因组所(国家生物信息中心)开发在单细胞中识别染色质类TAD结构的算法
基因组DNA和组蛋白以特定的形式高度折叠在细胞核中,这一高级结构即三维基因组学,对细胞核内的诸多生命活动至关重要。基于染色质构象捕获(3C),尤其是高通量技术(Hi-C,ChIA-PET)的发展极大推动了三维基因组的研究,发现了包括染色质拓扑相关结构域(TAD),染色质环等一系列层次化的结构特征。近年来,单细胞水平下的Hi-C研究成为了三维基因组的一个重要研究方向。单细胞Hi-C数据对深入理解染色质结构的动力学机制,建立高分辨率细胞发育图谱都具有关键意义。然而,单细胞Hi-C数据由于极度稀疏,目前主流的Hi-C数据分析算法对其无能为力,而针对单细胞Hi-C设计的算法亦表现不佳。因此,亟需新的计算方法来分析鉴定单细胞内的染色质高级结构。
7月27日,中科院北京基因组研究所(国家生物信息中心)张治华研究组开发了预测单细胞内类TAD结构的算法deTOKI,该研究以“DeTOKI identifies and characterizes the dynamics of chromatin TAD-like domains in a single cell”为题,发表在Genome Biology上。
该研究将deTOKI与适用于低分辨率水平Hi-C数据的新算法IS, deDoc, SpectralTAD和GRiNCH,以及先由单细胞Hi-C实验数据通过预测出高分辨率数据,再由已有算法鉴定类TAD域结构的新算法scHiCluster及Higashi六个软件进行综合比较,发现用deTOKI分析单细胞Hi-C数据结果优于其他六个软件。比较的内容主要基于两点,首先是将高分辨率水平的Hi-C数据进行下采样,比较下采样数据和原始数据中鉴定的类TAD域结构的相似度,然后对染色质结构进行三维建模,对各个模型分别生成高分辨率水平和单细胞水平的模拟Hi-C数据,比较两个数据中鉴定的类TAD域结构的相似度。文章随后还在已有的单细胞Hi-C实验数据上使用模块系数和结构熵等指标来评价软件的表现,deTOKI都优于其它算法。
新算法deTOKI有助于未来的单细胞内染色质高级结构的研究,基于deTOKI算法,研究发现了单细胞内的类TAD域结构和细胞类型的关系,以及其与组蛋白修饰、DNA甲基化等多组学数据的关联。该项研究丰富了对基因组结构和功能关系的认识,为三维基因组学研究提供了新思路。
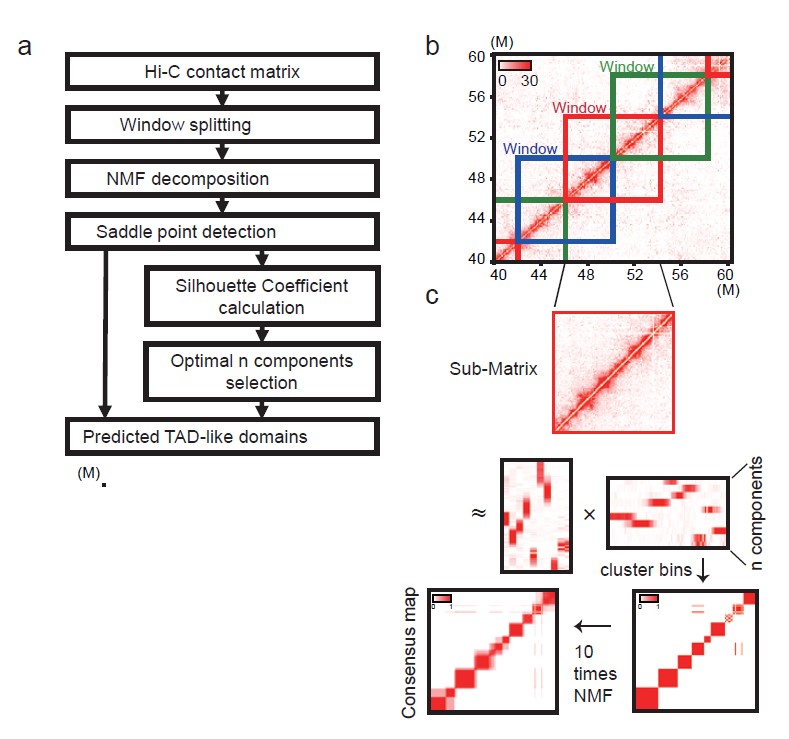
deTOKI的算法流程
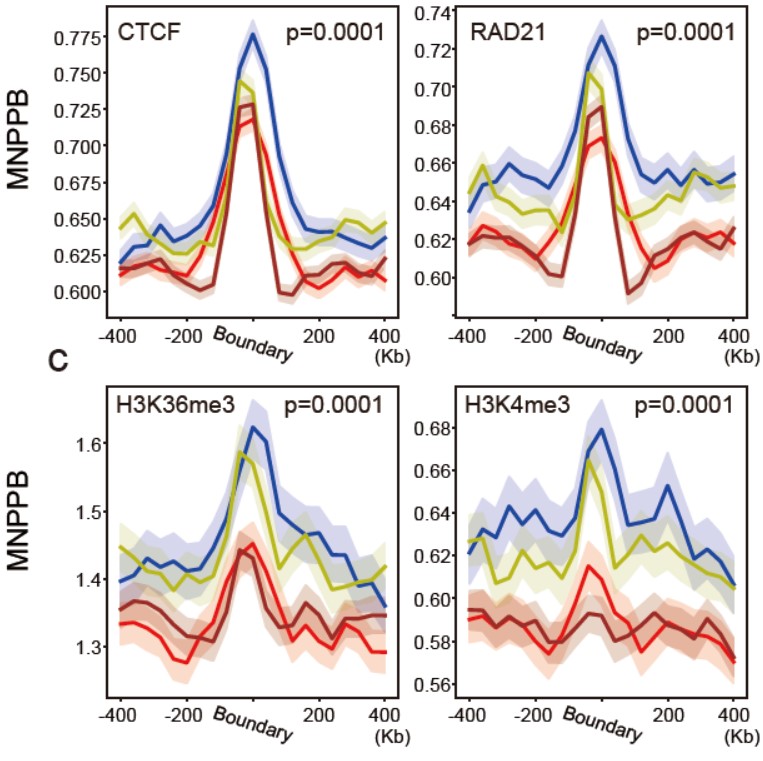
单细胞水平类TAD边界的表观修饰分布






