GSA:组学原始数据库系统
生命科学的发展已进入组学大数据时代,然而中国至今尚未形成可服务于科学研究的公共数据库存储体系。为了弥补这一空白,中国科学院北京基因组研究所生命与健康大数据中心开发并构建了组学原始数据存储归档系统Genome Sequence Archive(简称GSA;http://bigd.big.ac.cn/gsa 或 http://gsa.big.ac.cn)。GSA的系统建设遵循了国际核酸序列共享联盟(International Nucleotide Sequence Database Collaboration, INSDC)的相关标准,并作为INSDC的补充,旨在减轻国际相关数据库数据存贮及数据传输的压力;立足中国,服务全球。
引言
第二代高通量测序技术革新推动了生命科学研究的纵深发展与应用,尤其在人口与健康领域,世界众多国家相继启动了大型研究计划,如美国的精准医学研究计划 [1]、英国万人基因组计划 [2]、冰岛人群基因组计划 [3]、中国精准医学研究计划 [4]等。这些研究计划都将产生大量的组学数据,从而导致了生命健康组学大数据的爆炸性增长。与此同时,数据存储、整合与挖掘、转化与应用将成为重要的技术问题与挑战 [5,6]。
国际上,美国、欧洲和日本于2005年建立了国际核酸序列共享联盟(INSDC)[7],包括NCBI [8]、EBI [9]和DDBJ [10]三大数据库系统,形成领域内数据存储和共享使用的标准,接收并存储来自全世界科学家提交的组学数据。然而,中国是一个生物资源大国,也是一个数据产出大国;迫于学术论文的发表及学术期刊的要求,中国的科学家需要将大量的数据跨过海底线缆,提交到国际数据库。但由于中国国际网络出口带宽的瓶颈问题,数据传输效率低下。以中国科学院北京基因组研究所的150Mbs出口带宽为例,向NCBI数据库递交1TB的数据需要花费2周以上的时间。当前,中国已经启动国家级的精准医学研究计划以及若干大型的具有地域特色的研究任务。可以预见,未来中国每年将产生数十PB的组学数据;这将为目前的数据传输、存储与共享提出新的挑战。
为了缓解上述困难和问题,中国科学院北京基因组研究所开发并构建了组学原始数据库系统GSA,专注于组学原始数据收集与整合,并提供免费的数据存储、共享与访问服务 [11]。GSA遵循国际INSDC的数据标准及数据库建设标准,可收集来自不同测序平台产出的数据,并存储序列数据及其对应的元数据信息,确保数据的完整性。GSA立足于中国,极大的方便了中国科学家的数据递交;同时,服务于全球,为全世界的科研领域共享并贡献数据。
数据库内容和使用
数据结构与模型
为了确保与INSDC数据库系统的兼容性,GSA遵循了INSDC数据库系统的数据标准和数据结构,并将数据分为四类,即项目信息(BioProject)、样本信息(BioSample)、实验信息(Experiment)和测序信息(Run);数据结构如图1所示。
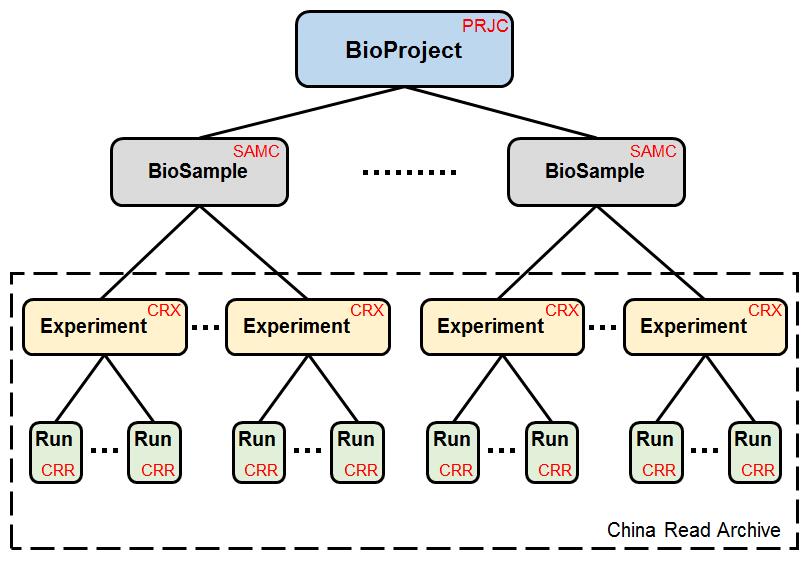
图1 GSA数据模型
项目信息的数据获取号(Accession Number)以“PRJCA”为前缀,其中字母“C”表示中国。项目信息提供了一个针对本研究任务的概要性描述,并包括研究目的、涉及的物种、数据类型、数据递交者、基金资助机构、发表的文章等信息。样本信息的数据获取号以“SAMC”为前缀,包含一些有关生物样本的描述信息如样本类型、样本属性等。实验信息以“CRX”为前缀,为特定样本实验处理方式,包括实验目的、文库构建方式、测序类型等信息。测序信息的数据获取号以“CRR”为前缀,内容主要包括测序文件和对应的校验信息。在四类数据中,项目信息和样本信息是独立运行的模块,而实验信息和测序信息形成了测序序列的归档库。基于上述标准和结构,GSA不仅方便数据递交,而且便于管理数据权限,实现数据共享与交换。
除此之外,GSA考虑大型项目管理的需求,引入Umbrella Project概念,提供大型合作型项目的伞装结构管理。目前,已有两个中国科学院战略先导项目和一个中国科学院重点研究项目正在使用GSA系统管理和共享项目数据。
数据归档与统计
GSA接收来自全球的数据递交,接收不同测序平台产出的组学数据,并支持通用的数据文件格式如FASTQ、BAM、VCF。同时,GSA对接收到的数据进行质量评估,确保数据的完整性和可用性。在数据安全方面,类似于INSDC数据库系统,GSA允许数据递交者设置其数据的访问权限(公开访问或受控限制);公开即意味着数据可被任何人访问或下载使用,受控即其他人的访问在一段时间内将被受到限制。在GSA系统后台,可被公开访问和受控访问的数据存储于不同的磁盘空间内,以确保数据的安全性。从2015年8月份GSA系统上线至今,系统中的数据呈现显著增长的趋势(图2),截止到2016年底,GSA已经接收了来自39个研究机构160余位科研人员的用户注册信息,并收录198个项目,8674个样本,9263个实验和10745个测序信息,涵盖了超过80个物种的信息。
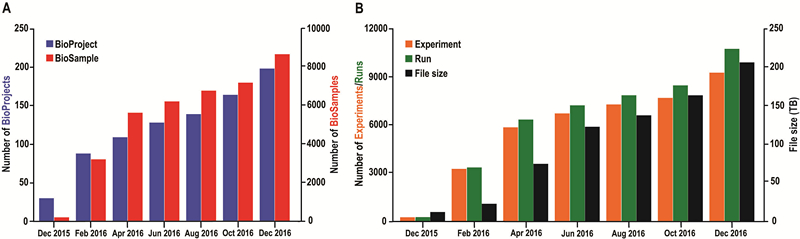
图2 GSA数据统计
数据递交与信息检索
GSA系统提供用户注册和登录功能,因此在创建一个数据递交前,首先需要通过GSA系统注册用户账户,在用户账号被验证通过并激活后,方可登录系统并创建数据递交页面。通常情况下,在GSA中完成一个数据递交需要执行五个操作,分别为注册项目、样本、实验、测序四类元数据信息和提交序列文件(图3)。在元数据信息收集页面,GSA系统提供友好访问的页面向导帮助用户实现信息录入;而针对测序文件上传,GSA提供基于IPV4和IPV6两条网络链路的FTP服务器,确保数据传输的高效性。GSA系统实现了数据全局检索功能,并对检索的结果进行分类统计;同时,用户可以预览检索出的每一个数据的详细信息。
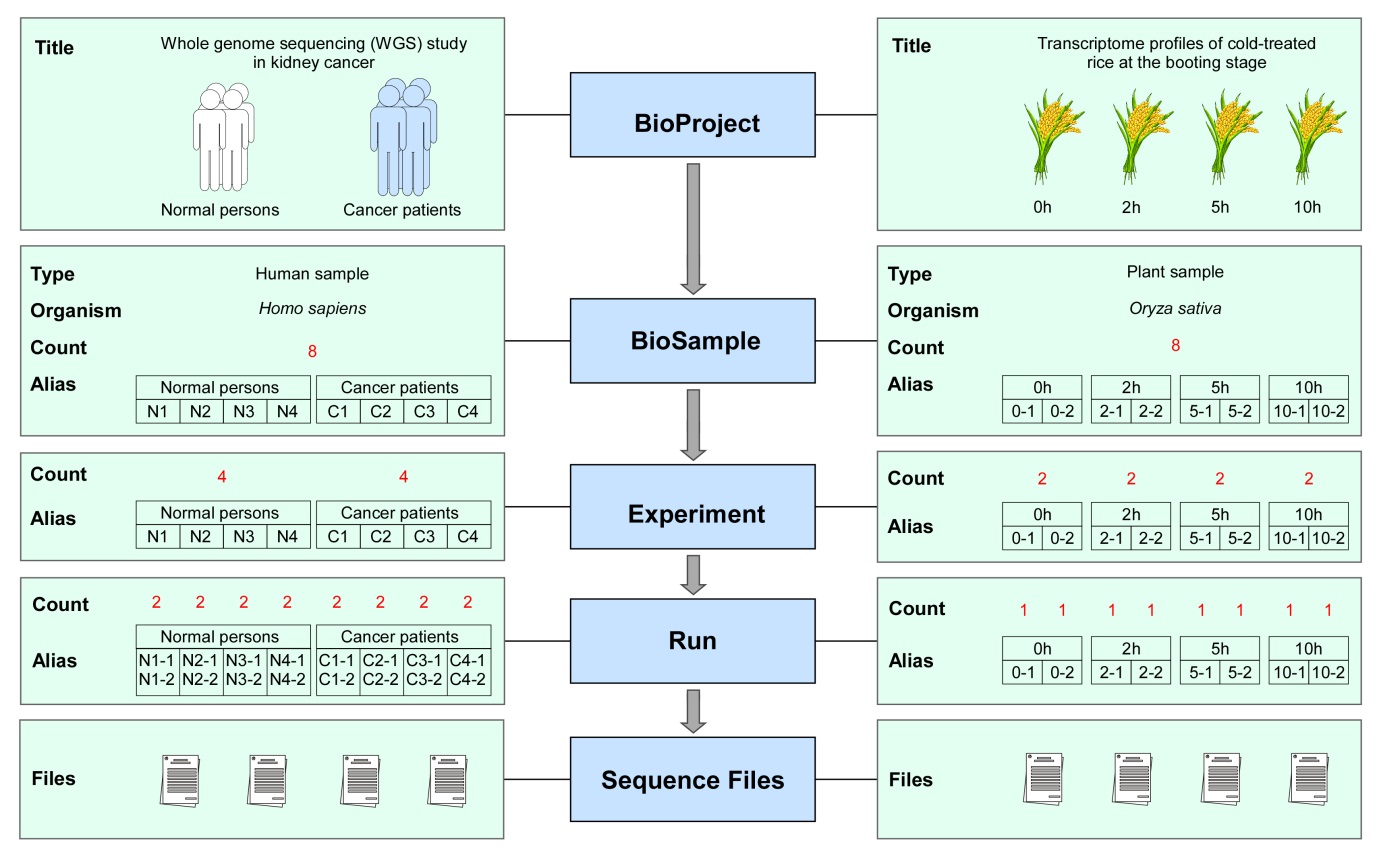
图3 GSA数据提交
发展与展望
俗话说“能力有多大,责任就有多大”。当今的中国已是世界第一大经济体,并在全球的经济体中发挥着越来越重要的作用。同样,在科研领域,在当今中国组学测序数据产量显著增加的情况下,我们应该承担起相应的责任,建立国际化的组学数据存储体系,分担国际数据库数据存储压力,服务于全球的生命科学研究机构。
GSA与国际同类数据库一样,致力于存储生命科学研究产出的组学大数据,并致力于中国组学数据汇交、管理、共享与应用体系的建设,促进中国在生命组学大数据领域的发展,提升中国在国际组学数据共享领域的地位,服务于全世界的生命科学研究与产业创新应用。 基于此,中国科学院北京基因组研究所发起“中国基因组数据共享倡议”(http://bigd.big.ac.cn/gdsd),呼吁中国产出的组学数据递交GSA进行统一存储、管理与共享。在倡议发出后很短的时间内,得到全国超过380个机构的1000余人支持本倡议。这代表了中国人的心声,也代表了中国众多科研资助机构的心声。
总结
GSA是一个公共的、免费的组学原始数据存储库,在建设标准上遵循国际INSDC数据库体系的数据标准和数据库结构标准,在内容上收集生命科学研究中产生的组学测序数据及其元数据信息,并且接受来自全世界科研人员的数据递交与获取请求。在组学大数据时代,GSA不仅作为当前INSDC数据库体系的补充以缓解组学大数据远距离传输与储存的压力,而且承担推动国际组学大数据共享的责任。
未来,GSA将逐步扩展与完善系统功能,提供专业化的组学大数据管理解决方案,如面向国家精准医学研究计划的组学大数据存储与管理,面向宏基因组数据的存储与管理等;另一方面将重点加强IT基础设施的建设,并提升数据存储能力和共享效率。
致谢
感谢罗静初教授和朱伟民教授给予GSA系统建设的诸多宝贵意见和建议。本项目也得到了国家项目基金的支持,主要有:中国科学院先导项目、国家高技术研究发展计划(863计划)、国家重点研究发展计划、中国科学院国际合作国际大科学计划、中国科学院重点部署项目、中国科学院关键技术人才项目。
参考文献
[1] Collins FS, Varmus H. A new initiative on precision medicine. N Engl J Med 2015;372:793–5.
[2] Taylor PN, Porcu E, Chew S, Campbell PJ, Traglia M, Brown SJ, et al. Whole-genome sequence-based analysis of thyroid function. Nat Commun 2015;6:5681.
[3] Gudbjartsson DF, Helgason H, Gudjonsson SA, Zink F, Oddson A, Gylfason A, et al. Large-scale whole-genome sequencing of the Icelandic population. Nat Genet 2015;47:435–44.
[4] Bai B, Zhao WM, Tang BX, Wang YQ, Wang L, Zhang Z, et al. DoGSD: the dog and wolf genome SNP database. Nucleic Acids Res 2015;43:D777–83.
[5] Xue Y, Lameijer EW, Ye K, Zhang K, Chang S, Wang X, et al. Precision medicine: what challenges are we facing? Genomics Proteomics Bioinformatics 2016;14:253–61.
[6] Zhang Z, Bajic VB, Yu J, Cheung KH, Townsend JP. Data integration in bioinformatics: current efforts and challenges. In: Mahdavi MA editor. Bioinformatics ––trends and methodologies. Rijeka: InTech;2011,p.41–56.
[7] Cochrane G, Karsch-Mizrachi I, Takagi T, International Nucleotide Sequence Database Collaboration. The International Nucleotide Sequence Database Collaboration. Nucleic Acids Res 2016;44:D48–50.
[8] NCBI Resource Coordinators. Database resources of the National Center for Biotechnology Information. Nucleic Acids Res 2016;44:D7–19.
[9] Cook CE, Bergman MT, Finn RD, Cochrane G, Birney E, Apweiler R. The European Bioinformatics Institute in 2016: data growth and integration. Nucleic Acids Res 2016;44:D20–6.
[10] Mashima J, Kodama Y, Kosuge T, Fujisawa T, Katayama T, Nagasaki H, et al. DNA data bank of Japan (DDBJ) progress report. Nucleic Acids Res 2016;44:D51–7.
[11] BIG Data Center Members. The BIG Data Center: from deposition to integration to translation. Nucleic Acids Res 2017;45:D18–24.
该文英文发表在Genomics, Proteomics & Bioinformatics期刊2017年第一期,全文链接http://www.sciencedirect.com/science/article/pii/S1672022917300025;http://gpb.big.ac.cn/






