基因组所抗癌药物分子机制研究获进展
近日,中国科学院北京基因组研究所雷红星研究组开展的“抗癌药物的分子机制研究”取得阶段性进展,其研究论文《Early stage intercalation of doxorubicin to DNA fragments observed in molecular dynamics binding simulations》,于2012年6月在《Journal of Molecular Graphics and Modelling》杂志发表。该文采用分子动力学的方法研究了阿霉素分子从自由状态到嵌入DNA碱基片段的动态过程,提出了一个新的药物分子插入机制(打开-插入机制),即插入过程是经外部结合后伴随着碱基对的打开(base-flipping)进行的,这一新机制的提出对于抗癌药物分子机制研究起到了积极的推动作用,为设计更有效的抗癌药物提供了理论依据。
阿霉素是一种临床上广泛使用的抗癌药物,它能有效治疗急性白血病,胃癌,肝癌等多种恶性肿瘤疾病。目前认为其作用机制是通过嵌入癌细胞的DNA碱基片段中,阻碍DNA的转录和复制,从而抑制肿瘤生长。但具体嵌入过程和分子机制还不是十分清楚。
为此,基因组所雷红星研究员及其科研团队,从未结合的自由态(一段B型DNA片段和连个自有的阿霉素分子)出发进行了全原子的分子动力学模拟。整个模拟过程验证了力场的可靠性,并以较高的空间和时间分辨率刻画出了结合过程中的结构和能量的动态变化规律。从模拟过程的轨迹中研究人员观测到阿霉素分子与DNA结合的三种模式,包括一端结合、DNA小沟结合以及碱基之间结合。其中,结合到DNA小沟中是到最终插入状态的一个中间态,即“外部结合态”;结合到碱基之间要经历碱基对被打开(base-flipping)的过程。这种打开-插入的机制与之前提出的碱基对之间距离拉开再嵌入的机制有很大不同。拉开-插入机制需要使碱基对之间产生较大空间才可使药物分子插入,而打开-插入机制却不需要。
论文链接:http://www.sciencedirect.com/science/article/pii/S1093326312000678?v=s5
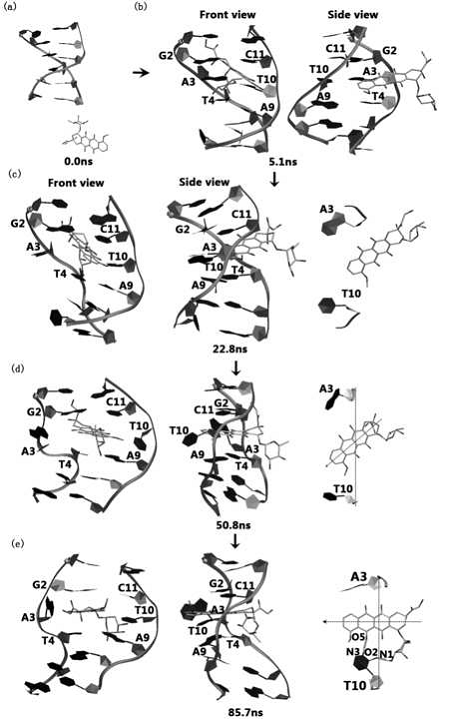
图:d(CGATCG)2序列插入轨迹的代表结构






