国家生物信息中心合作揭示RNA聚合酶II如何精准解码基因组中的“剪接密码”
基因组就像一本“生命天书”,而mRNA剪接是解读这本天书的关键一环。在人类基因组中,超过95%的基因都含有内含子,这些非编码序列必须被准确剪除,才能生成具有功能的成熟mRNA。然而,基因组中存在大量“假剪接位点”(pseudo splice sites),它们在序列上与真实的剪接位点高度相似,却并不具备真正的剪接功能。如何在共转录(co-transcriptional)过程中精准识别真实剪接位点,是维持基因表达精确性的关键。
9月26日,国家生物信息中心计算生物学部郝亚静团队与西湖大学付向东团队合作,首次揭示了RNA聚合酶II(Pol II)作为唯一能在转录过程中“线性扫描”剪接位点的分子机器,如何通过“U2AF蛋白的动态循环”机制,在转录过程中精准识别真实的剪接位点的分子机制。该成果以“Dynamic U2AF cycling defines two phases of cotranscriptional pre-mRNA splicing”为题, 发表在《科学》(Science)期刊,为理解基因表达调控提供了全新的视角。
Pol II:基因组信息的“精准解码器”
Pol II不仅是转录的核心引擎,更是共转录剪接过程中的关键调控者。本研究的一项重要发现挑战了长期以来“RNA聚合酶II的CTD结构域对招募剪接因子至关重要”的传统观点。然而,迄今为止,CTD 在招募剪接因子中的作用尚未被直接验证。通过使用CTD缺失的Pol II变体,研究人员证实Pol II可以在不依赖CTD的情况下与U2AF1、SNRNP70等剪接因子结合,揭示了一种独立于CTD的共转录剪接调控机制。
该研究首次系统揭示了Pol II如何在转录过程中动态调控剪接因子的招募与释放,实现对剪接位点的精准识别:Pol II通过其亚基RPB9直接结合U2AF1,识别新生RNA中的3’剪接位点(AG二核苷酸);同时,相关报道证实其亚基RPB2和RPB12结合U1 snRNP,识别5’剪接位点(GU二核苷酸)。这使得Pol II能够在转录过程中对成对的5’和3’剪接位点进行实时的线性扫描,从而在源头上避免大量假剪接位点的错误识别,确保剪接的准确性与基因表达的精确调控。
U2AF1单体与Pol II的特异结合:打破传统认知
传统观点认为,U2AF1与U2AF2总是以稳定异源二聚体(heterodimer)形式存在,并共同参与剪接。然而,本研究首次发现(1)U2AF1以单体的形式直接结合在Pol II上;(2)而U2AF2以单体形式加入后,与U2AF1结合形成异源二聚体,进而触发剪接初始复合物从Pol II上释放,进入后续剪接体组装催化阶段。这一发现打破了“U2AF1必须与U2AF2结合共同发挥作用”的传统认知,揭示了U2AF蛋白在剪接过程中动态循环的新机制。
U2AF动态循环模型:定义共转录剪接的两个阶段
基于上述发现,研究团队提出了“U2AF动态循环”模型(Dynamic U2AF Cycling),将共转录剪接划分为两个阶段:(1)Pol II依赖的剪接位点识别阶段:Pol II通过RPB9招募U2AF1单体,RPB2和RPB2结合U1 snRNP,实现对新生RNA中成对剪接位点的识别,防止“假剪接位点”的错误识别。(2)Pol II非依赖的剪接体组装催化阶段:U2AF2与U2AF1结合形成异源二聚体,进而触发剪接初始复合物从Pol II上释放,剪接体进入后续组装和催化阶段,不再依赖Pol II。伴随着Pol II的持续转录和延伸,释放后的Pol II可以重新招募新的U2AF1单体和U1 snRNP,开启对下游剪接位点的成对识别,从而实现剪接过程的高效、精准、连续进行。
西湖大学邵长伟和国家生物信息中心郝亚静研究员为本文的共同第一作者,西湖大学付向东教授为本文通讯作者。
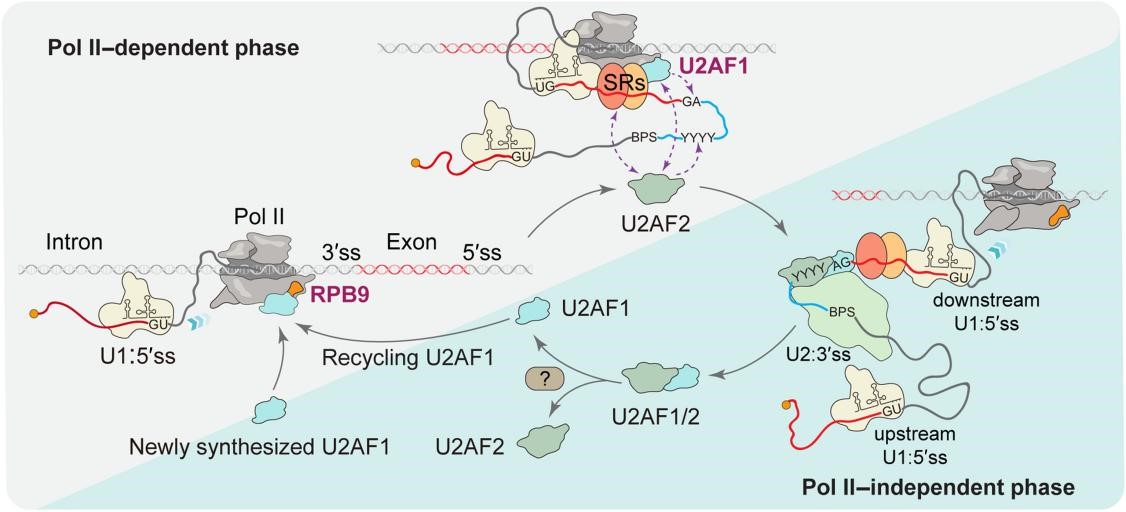
U2AF周期调控两个共转录剪接阶段示意图






