国家生物信息中心合作研发基因编辑动物新发突变数据库VDGE
基因编辑动物在生物科学和医学研究中发挥着至关重要的作用,并且在农业领域展现出巨大的实际应用潜力。随着基因编辑技术的迅猛发展,基因编辑动物的数量在过去十年间急速增长。科学界越来越关注基因编辑系统对生物基因组的影响及基因编辑动物中可能出现的新发突变问题。然而,目前仍然缺乏对基因编辑动物数据的整理、汇总以及标准化分析,限制了研究人员深入挖掘和利用这些数据。
10月29日,国家生物信息中心赵文明团队、昆明动物研究所王国栋研究组、遗传与发育生物学研究所张永清研究组联合,以“VDGE: a data repository of variation database for gene-edited animals across multiple species为题在Nucleic Acids Research 期刊发表论文,从实验室的基因编辑猴和基因编辑犬出发,同时收集、整理并分析了目前具有家系全基因组数据的相关数据集,构建了基因编辑动物新发突变数据库VDGE,首次实现了对基因编辑动物新发突变的标准化分析、整合和展示,为相关数据的深入挖掘和充分利用提供了一个综合的信息平台。
VDGE包括6个关键模块,分别是物种(Species),家系(Animal trios),样本(Samples),目标突变(On-target mutations),以及变异(Variations)和相关基因(Genes)。VDGE目前的数据主体来源于具有家系全基因组测序数据的相关数据集,涵盖了恒河猴、食蟹猴和犬等物种的107个动物家系,174个全基因组测序样本,56个目标突变,115,710个变异及12,708个相关基因。除此之外,VDGE还整合了部分缺乏全基因组测序数据的基因编辑猪和犬,以及它们的相关目标突变信息。未来,VDGE将进一步整合更多的基因编辑物种及多种类型的变异数据,为领域内的研究人员提供更加全面和多样化的数据资源平台。
国家生物信息中心赵文明正高级工程师、昆明动物研究所王国栋研究员、遗传与发育生物学研究所张永清研究员为该论文的共同通讯作者。遗传与发育生物学研究所工作人员石文文、国家生物信息中心博士生金恩慧、遗传与发育生物学研究所数据中心方璐为该论文的共同第一作者。该研究得到科技创新2030项目、国家重点研发计划、中科院先导专项和春城计划等基金项目资助。
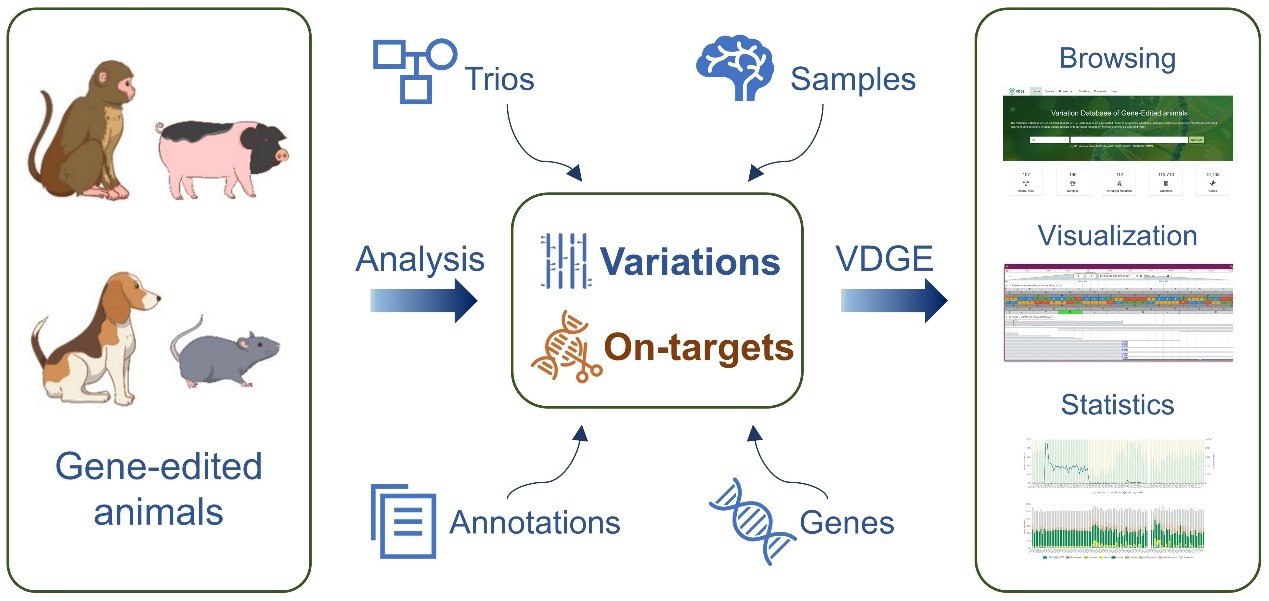
VDGE数据库构建流程及内容展示






