国家生物信息中心合作揭示并校正多组学数据中的细胞周期干扰效应
在多组学(Multi-Omics)研究日益成为解析生命过程重要手段的今天,我们是否真正捕捉到了数据背后的“生物学本质”?细胞周期,这一在经典生物学中早已被广泛认知的基本过程,在多组学分析中却往往被当作背景因素忽略。然而,细胞周期的异质性,尤其是不同细胞类型间细胞周期结构的差异,干扰了我们对组学数据的解读,导致差异信号的伪阳性,掩盖了真正具有生物学意义的发现。
5月28日,国家生物信息中心蔡军团队联合中国农业科学院北京牧医所郑彩宏研究员团队,在Advanced Science 在线发表了题为“Mitigating Cell Cycle Effects in Multi-Omics Data: Solutions and Analytical Frameworks”的研究论文。该研究以干细胞与分化细胞为模型,系统梳理了细胞周期结构差异对多组学数据解释的全局性影响,构建了一套针对不同组学类型的通用分析框架,为如何鉴定高增殖细胞群体中真实的生物学信号提供了科学依据和技术路径。
在干细胞研究、胚胎发育及肿瘤等增殖活跃的生物学场景中,细胞周期结构的差异是组学数据解读偏差的重要来源之一。研究指出,当样本中处于S期的细胞比例超过38%时,DNA的非同步复制会影响拷贝数变异的识别,导致高假阳性率。对此,研究团队引入复制时序域(Replication Timing Domain,RTD)校正,可以显著降低拷贝数变异鉴定的假阳性。
不仅如此,细胞周期也影响了染色质可及性和DNA甲基化的识别精度。研究发现,S期特异的染色质开放区域(open chromatin regions,OCRs)更富集于早期RTD区域,并且与OCRs关联的假阳性拷贝数变异区域倾向于更高比例拷贝数扩增,同时,细胞周期不同阶段共有的OCRs,往往具有更高的转录活性关联性与调控功能。此外,差异甲基化区域的识别准确性也受到细胞周期的影响。通过整合转录组数据,研究进一步表明,相较于直接比较,基于细胞周期分型后进行独立比较的策略,更有助于揭示潜在的生物学信号。 在单细胞转录组层面,尽管目前已有多种单细胞分析方法试图消除细胞周期的干扰,但这些方法往往不足以彻底剥离细胞周期变量对差异表达基因识别的影响。通过对多种细胞周期分类工具的比较,研究最终选定了分类准确性较高的Cyclone算法,并在此基础上构建“phase comparison”策略,有效提升差异表达基因的生物学相关性和功能注释精度。
基于此,研究团队系统评估了细胞周期在多组学分析中的干扰效应,并针对不同数据类型提出了解决策略,主要包括:通过RTD校正降低高S期比例群体中的CNV假阳性,优化甲基化与转录组分析流程。结合Cyclone算法与“phase comparison”方法,有效提升单细胞数据中差异基因的识别准确性。团队通过模拟数据、生物学实验和功能注释等多维手段,系统验证了策略的有效性。
综上所述,研究团队揭示了细胞周期这一“被忽略的重要变量”,在多组学解读中的关键作用。通过将细胞周期明确纳入分析流程,提升了组学数据的解释准确性,为干细胞及肿瘤等高增殖背景下的精准分析提供了研究范式。
该研究得到了北京市自然科学基金重点研究专题课题、中国科学院关键核心技术攻坚先导专项、中国科学院前瞻战略科技先导专项等项目资助。国家生物信息中心蔡军研究员、中国农业科学院北京牧医所郑彩宏研究员为本文的共同通讯作者。国家生物信息中心博士生聂瑞与北京牧医所郑彩宏研究员为本文的共同第一作者。
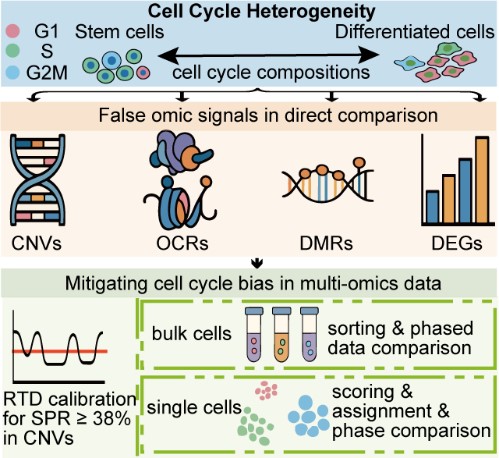
细胞周期对多组学影响及校正示意图






