北京基因组所合作开发单细胞测序数据中细胞子群特异DNA甲基化鉴定新方法
中国科学院北京基因组所吕雪梅研究组与美国弗吉尼亚理工大学谢荷煌研究团队合作,利用小鼠胚胎干细胞得到的单细胞DNA甲基化测序数据,开发了一套生物信息分析流程,挖掘ESCs中的细胞子群特异DNA甲基化(CSM)。相关成果以“Integrative single-cell omics analyses reveal epigenetic heterogeneity in mouse embryonic stem cells”为题在线发表于PLoS computational biology杂志。
由于当前单细胞甲基化组数据具有覆盖细胞数少、测序乘数低以及DNA片段扩增偏好等缺陷,导致对等位基因特异甲基化(ASM)、非对称性甲基化(AM)在内的细胞内DNA甲基化异质性,和真正的CSM之间的区分能力十分受限。因此科研人员在过滤掉这些细胞内甲基化变异的干扰后,开发了新的统计模型,即“混合贝塔模型”以鉴定出真正的CSM位点。
该研究进一步整合了胚胎干细胞单细胞转录组数据、小鼠成体组织甲基化组数据和转录因子结合基序注释信息,对CSM的调控模式进行解读。结果发现CSM位点显著富集到CGI shelf区域,以及增强子和启动子偏好的组蛋白修饰区域;同时CSM定位到的基因在单细胞间表现出广泛的表达差异。进一步研究发现,CSM位点可以被聚类成共甲基化的模块,这些CSM模块在不同成体组织中具有不同的甲基化水平分布,并且表现出模块特异的转录因子富集。该方法为单细胞甲基化组测序数据表观异质性的深度挖掘提供了方法学的创新,对于研究复杂细胞群体异质性具有重要指导意义。
该研究得到国家自然科学基金、中国科学院战略性先导科技专项等基金及动物进化与遗传前沿交叉卓越中心的资助。
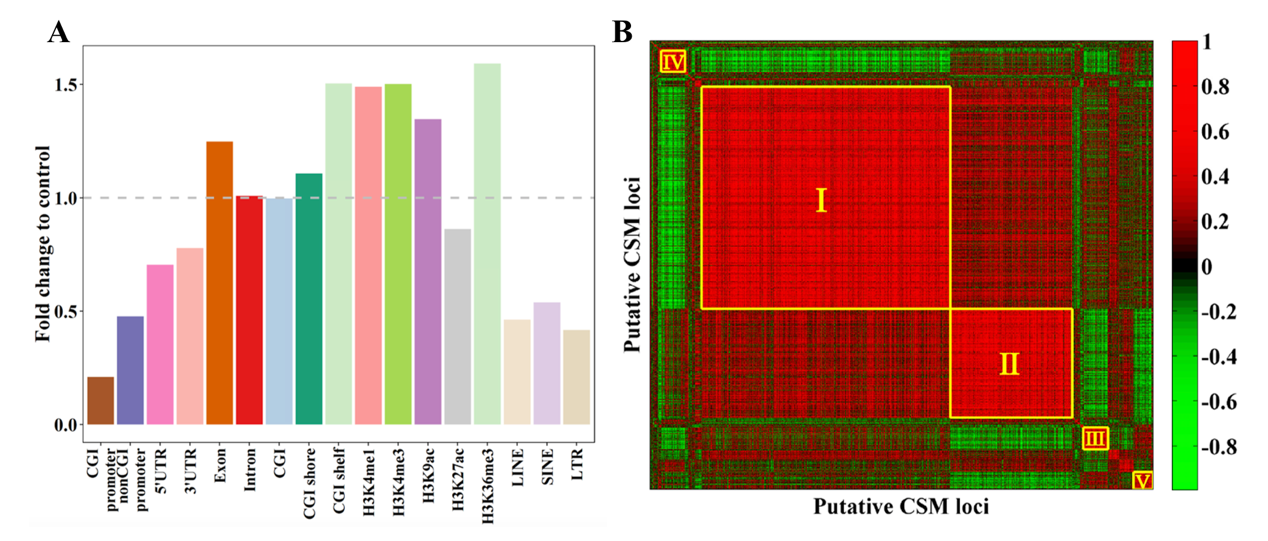
(A) CSM位点在不同基因组特征区域上的富集分布;(B) 两两CSM位点的皮尔森相关系数热图,前五
共甲基化CSM模块按罗马数字序标注;颜色从绿到红表示相关系数从-1到1






