近日,基因组所在国际生物多样性与健康大数据(Global Biodiversity & Health Big Data,简称BHBD)联盟框架下,与沙特阿卜杜拉国王科技大学、巴基斯坦真纳大学以及美国梅奥医学院开展科研合作,联合开发了可跨物种使用的长非编码RNA(long non-coding RNA, lncRNA)鉴定软件LGC。该成果以“Characterization and identification of long non-coding RNAs based on feature relationship”为题在国际学术期刊Bioinformatics在线发表。
测序技术的飞速发展产生了大量转录组数据,而转录组分析中一个重要的研究内容是鉴定新的转录本并且检测其蛋白编码能力。虽然利用蛋白质谱检测、Ribo-seq等实验方法可以获得蛋白编码证据,但成本高、耗时长且通量低。因此,目前主要依赖软件预测的计算方法鉴定lncRNA。已有相关软件大多需要高质量的训练集,对于缺乏高质量数据的非模式物种,鉴定其lncRNA难度极大,亟需发展支持跨物种使用的lncRNA准确鉴定方法与工具。
为解决以上难题,北京基因组所联合多家科研机构,研究了蛋白编码RNA与lncRNA在多物种中普遍存在的序列特征差异,发现GC含量与ORF(开放读码框)长度的关系在两者间存在显著差异,且这种差异在动植物中广泛存在。基于此,研发了能够跨物种使用的lncRNA鉴定方法和软件LGC。该方法不需要物种特异训练集,与同类方法相比,LGC可实现从植物到哺乳动物的lncRNA的准确鉴定,且具有较为平衡的灵敏度与特异性,为lncRNA跨物种鉴定以及功能分析提供重要方法和工具。同时,提供web server,支持fasta、gtf、bed三种格式,实现在线分析计算。Fasta格式不限定物种,gtf与bed格式目前支持人、小鼠、斑马鱼与果蝇的在线分析。
该项研究得到了中国科学院战略性先导科技专项、国家重点研发计划、中国科学院“十三五”信息化专项等基金的资助。
Web server
文章链接
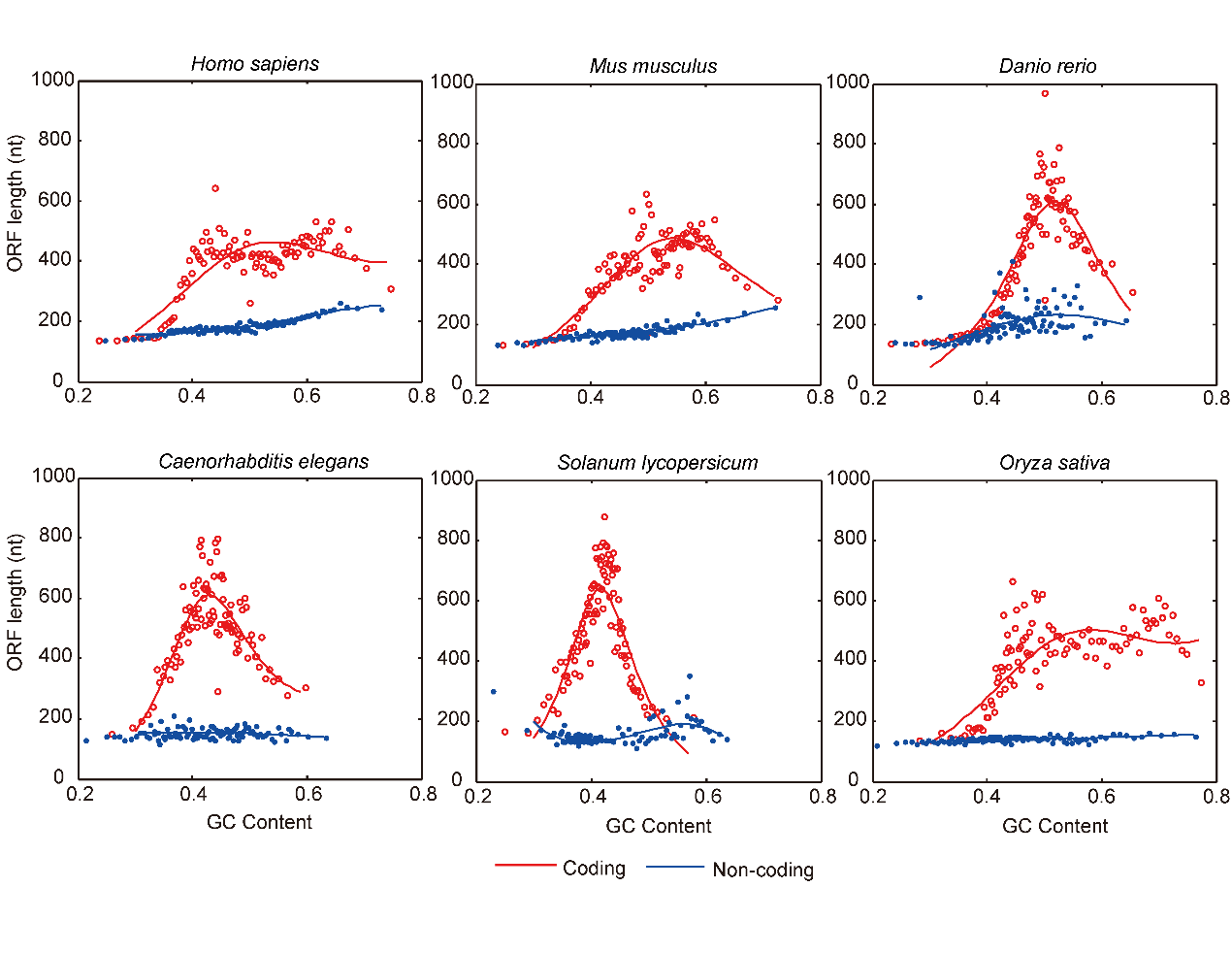
GC含量与ORF长度的关系在蛋白编码RNA与lncRNA中存在显著差异


