一、中心成立及定位
中国科学院北京基因组研究所于2016年2月29日成立生命与健康大数据中心,是研究所三大科研体系之一。中心面向我国人口健康和社会可持续发展的重大战略需求,围绕国家精准医学和重要战略生物资源的组学数据,建立海量生物组学大数据汇交、存储与管理的应用与共享平台,发展组学大数据系统整合、挖掘与分析的新技术、新方法。2019年6月5日,由科技部、财政部批复,“国家基因组科学数据中心”依托中国科学院北京基因组研究所生命与健康大数据中心,联合中国科学院上海生命科学研究院和中国科学院生物物理研究所共同建设。

定位与目标
二、中心运行机制与组成
中心主任:鲍一明研究员
中心常务副主任:章张研究员
中心副主任:赵文明高级工程师
工作团队:组学原始数据归档库、基因组数据序列库、基因组变异数据库、基因表达数据库、表观基因组数据库、生命科学维基知识库、中国人群参比数据库、电子健康管理系统和系统运维部等九个工作团队

组织与结构

大数据中心集体照
三、年度主要科研进展
1. 进一步完善国内从数据汇交存储、整合挖掘到转化应用的数据库体系
国家基因组科学数据中心自2019年6月正式成立以来,在依托单位中科院北京基因组研究所,和共建单位中科院生物物理所和上海营养健康所前期数据资源建设基础之上,从数据、信息、知识三个层次,系统推进面向人口健康和重要战略生物资源的生物大数据汇交共享平台和多层次资源体系建设。2019年,国家基因组科学数据中心发布了基因组变异与表型关联、微生物分类与基因组资源、表观组关联分析、特色物种多维组学信息资源等8个新开发数据库,系统更新了原始测序数据归档库GSA、基因组归档数据库GWH、基因组变异数据库GVM、非编码RNA等15个资源库,并对云分析等4个网络服务工具进行了更新优化。
国家基因组科学数据中心免费向国内外用户提供方便快捷的多组学数据汇交和存储服务,目前,已汇交来自269个单位720个用户递交的超过1.4PB的组学数据,相关数据发表于144种国内外期刊的237篇文章。GSA已被国际著名出版商Elsevier收录为指定的基因数据归档库。此外,通过跨库检索形式,中心还整合了13家国内合作科研机构的25个专业特色数据库,极大丰富了数据资源类型。
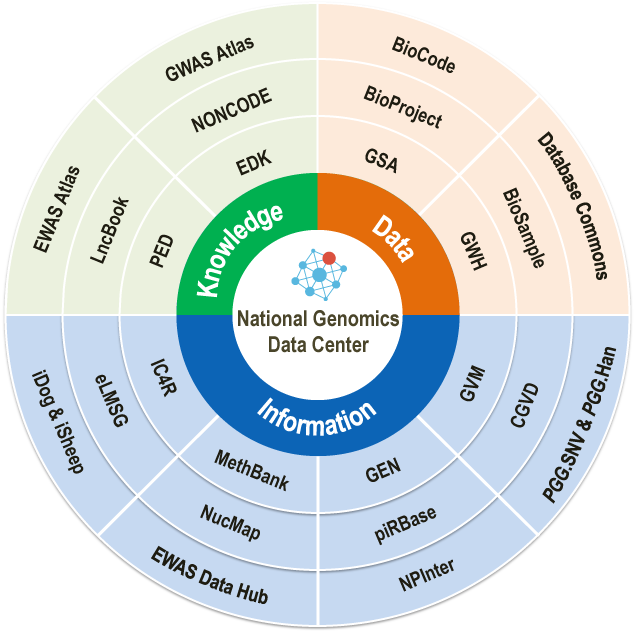
国家基因组科学数据中心核心数据资源 (Nucleic Acids Res, 2019)
2. 开发基因组变异与表型关联知识库:GWAS Atlas
全基因组关联分析(GWAS)在全基因组范围内筛选出与表型性状等相关联的遗传位点,是挖掘生物复杂性状遗传基础的关键技术。随着测序技术和分析算法的快速发展,越来越多GWAS研究工作被成功开展,并已解析了与许多动植物复杂性状相关联的遗传位点。但这些知识信息都分散在不同文献中,不利于知识整合、挖掘与再利用。本研究通过关键词检索、人工审编、词条比对注释等技术手段,结构化整理了现有主要农作物和畜牧动物的基因型-表型关联知识,开发了世界上首个动植物基因组变异-表型关联知识库GWAS Atlas。
GWAS Atlas知识库整合了9个物种(包括棉花、梅花、玉米、油菜籽、水稻、高粱、大豆等7种植物和山羊、猪等两种动物)与614个性状关联的75467条基因型-表型(G2P)信息,并通过语义比对等映射到五个不同的性状本体上(植物性状本体PTO,家畜性状本体ATOL,作物本体CO等),方便用户通过基于本体的层级结构来查找感兴趣的性状及对应的G2P关联信息。此外,研究人员还分析并定义了与多个性状相关联的多效基因及遗传位点,支持用户通过不同模块在线浏览、检索与下载。
GWAS Atlas 是全基因组变异信息数据库(Genome Variation Map,GVM)的延伸,将为未来重要农艺性状的模块化遗传研究和育种应用提供重要资源和平台。该项研究成果以“GWAS Atlas: a curated resource of genome-wide variant-trait associations in plants and animals”为题在国际学术期刊Nucleic Acids Research在线发表。
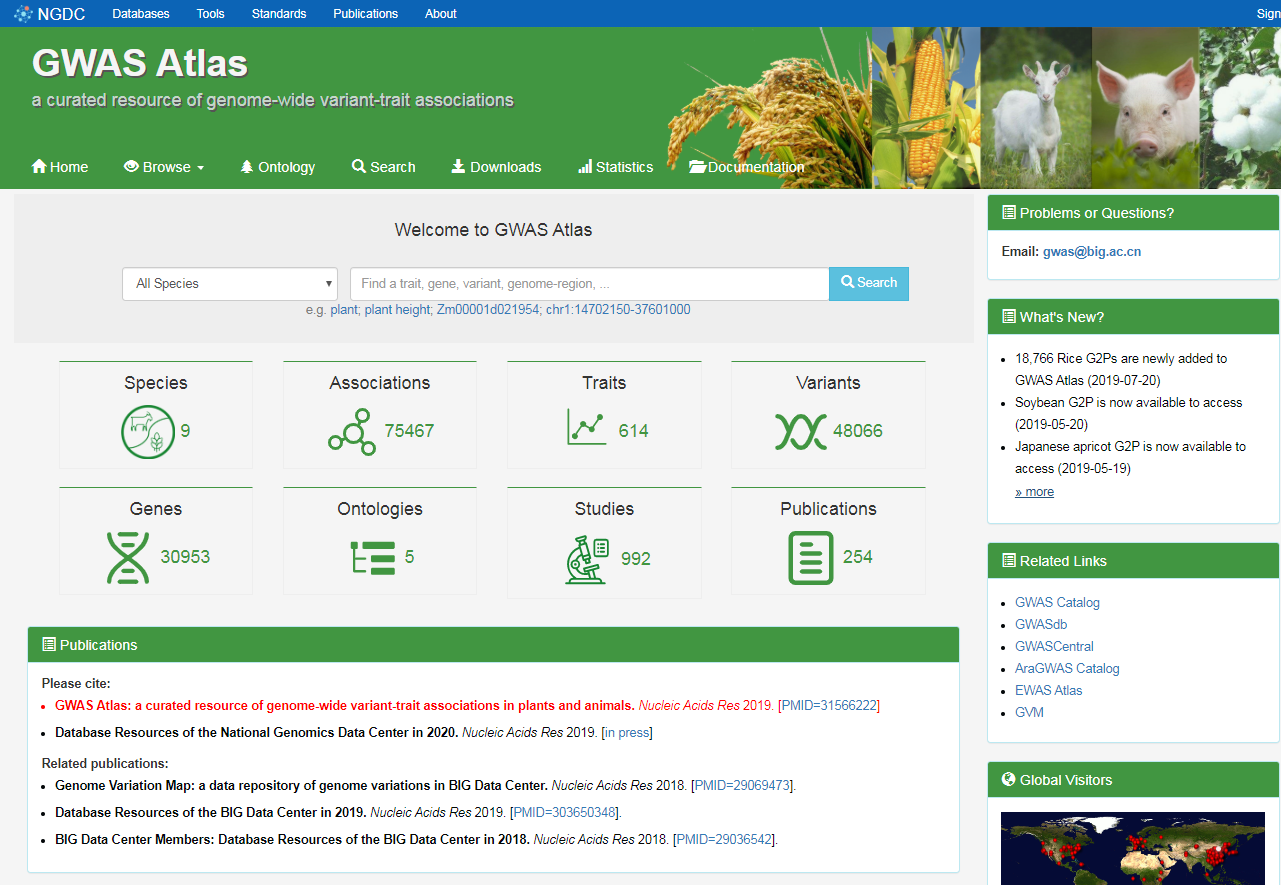
GWAS Atlas主页
3. 表观关联分析数据库系统构建
近年来,表观关联分析(EWAS)已成为探索复杂性状表观遗传基础的有效策略。DNA甲基化芯片数据和元数据的全面整合对于系统地表征和研究不同实验条件下的甲基化状态以及探索与各种性状相关的表观遗传机制具有根本意义。中心开发了针对EWAS的人工审编知识库EWAS Atlas,以及针对EWAS的DNA甲基化芯片数据存储和分析的数据库EWAS Data Hub,构建了较为完整的表观遗传数据库系统。
EWAS Atlas是一个完全基于人工审编和文献挖掘的知识库。当前版本的EWAS Atlas主要关注DNA甲基化这一重要的表观修饰,EWAS Atlas一共整合了618篇文献中1,038个研究报道的472,268个高质量的甲基化与表型关联。这些关联一共涉及到140个组织/细胞,2,786个队列以及419种表型本体。此外,EWAS Atlas还配备了功能强大的表型富集工具,用于研究表型与表型、表型与表观变异的关系。
EWAS Data Hub整合了来自NCBI、TCGA、EBI和ENCODE的75,344个样本的DNA甲基化芯片数据和对应的元信息,并采用了有效的归一化方法来消除不同数据集之间的批次效应。EWAS Data Hub为485,512探针和36,397基因,提供了一系列相关的评估值(包括组织特异性,年龄相关性,性别差异和种族特异性)和不同背景下的参考DNA甲基化图谱,涉及81种组织/细胞类型(包含25个脑部和25种血细胞类型),67种疾病(包括39种癌症),年龄,性别,种族和BMI。
表观遗传数据库系统的构建对于系统地表征和研究不同实验条件下的甲基化状态以及探索与各种性状相关的表观遗传机制具有根本性意义。
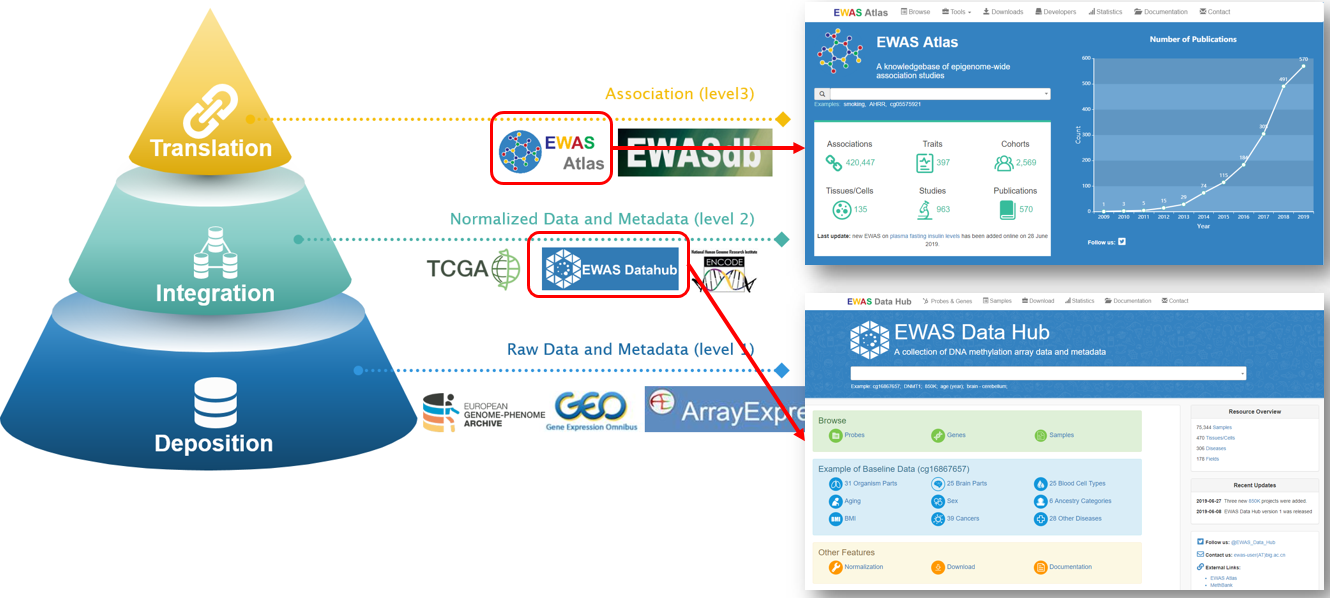
表观遗传数据库系统
4. 建立原核生物防御系统基因数据库
原核生物防御系统基因数据库(Prokaryotic Antiviral Defense System, PADS),收集、整合分析6,600,264个防御系统相关基因,这些基因来自古细菌和细菌的33,390个物种的63,701个基因组,分属于18个不同的防御系统(图1A)。PADS整合了防御基因注释和演化分析功能,同时还通过泛基因组学分析,将防御基因动态变化信息可视化展示。在浏览模块中,所有完成图基因组通过不同的分类学层次可视化展示。在搜索模块中,用户可以通过四种搜索方法查询所需信息,主要包括防御系统类别、防御系统子类型和基因名等。在分析模块中,PADS集成一套防御系统基因在线交互注释分析流程,综合序列同源性搜索、多序列比对、系统发育分析等功能。另外,基因保守度是理解防御系统机制的重要特征。为可视化防御系统相关基因跨物种的动态变化,PADS还集成了基因存在缺失变异(Presence–Absence Variation,PAV)分析功能。在PAV分析中,用户可以选择一个感兴趣的物种来查看PAV分析结果(图1B)。同时,用户也可以基于泛基因组分析结果,选择一个防御系统来查看防御系统相关基因在物种水平上的动态变化。PADS是一个开放的防御系统基因综合性数据库,可以有效促进原核生物防御系统研究并为分子工具开发提供参考信息。

原核生物防御系统基因数据库(A)数据库首页信息 (B)PAV分析热图
5. 研发家犬多组学综合性数据库系统:iDOG
中心与昆明动物研究所合作,通过整合挖掘公共数据和自产数据,形成集基因组、变异组、表观组、转录组等多组学为一体的综合性组学数据资源库。该项研究成果以“iDog:an integrated resource for domestic dogs and wild animals”为题在国际学术期刊Nucleic Acids Research在线发表。iDog是第一个致力于家犬(Canis lupus familiaris)和野生犬科动物的综合性数据资源库,为全世界从事犬科研究的科研人员提供各种数据服务和在线分析工具,同时也为全世界的养狗爱好者提供家犬品种、疾病等信息查询平台。
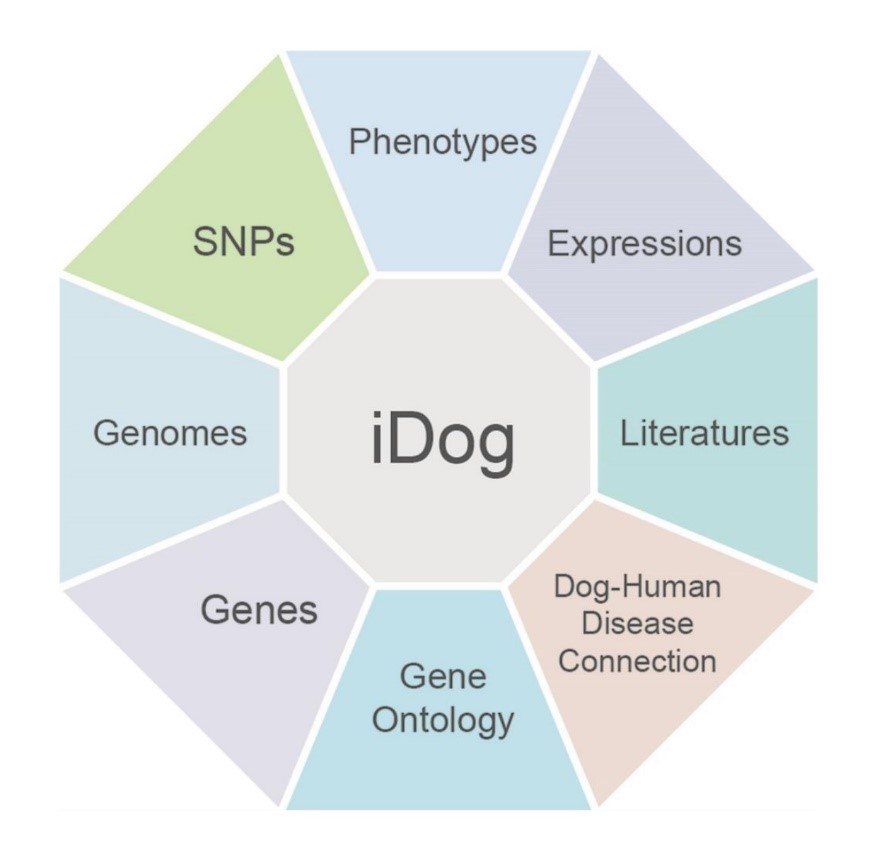
家犬多组学数据资源构成
6. 研发生物进化与多组学综合分析软件云平台:eGPSCloud
中心与中国科学院计算生物学重点实验室、北京生命科学研究院等多家单位组成联合攻关团队,合作开发的生物进化与多组学综合分析软件eGPS 1.0正式在线发布。该项研究成果以“EGPS 1.0: Comprehensive software for multi-omic and evolutionary analyses”为题于2019年6月18日在线发表于National Science Review。eGPS1.0收集了生物进化与多组学分析领域的主要软件与可视化工具,为全世界从事生物进化与多组学分析的科研人员提供免费的分析平台。
eGPS软件包括单机软件版本eGPS Desktop和云计算eGPS Cloud,将基因组分析、群体数据分析、进化数据分析、网络分析以及图形可视化这五部分的分析有机整合起来,实现远程云计算功能,方便用户在缺乏计算资源的情况下快速获得运算结果,并最终以图形、图表等形式直观展示。在eGPS Cloud和其他开放式在线资源的支持下,eGPS Desktop提供了一键点击从候选基因到基因树的分析流程。egps结合了云计算和桌面应用的优势,具有用户友好的图形界面和高度的交互能。
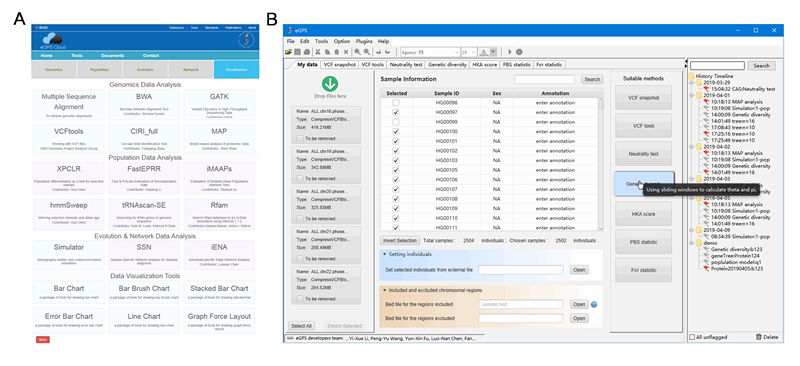
(A) eGPS Cloud网页界面,共包括15个软件以及20个可视化工具。(B) eGPS Desktop软件界面,共包括3大类16个功能模块,并且支持第三方插件
7. 成功举办第四届国际生命与健康大数据论坛
10月13日至16日,第四届国际生命与健康大数据论坛(The 4th Big Data Forum for Life and Health Sciences)在中国科学院北京基因组研究所成功召开。本次论坛依托中国科学院“国际健康大数据共享计划”,由北京基因组所国家基因组科学数据中心和中国遗传学会联合主办,共有来自国内外数十家单位的200余名代表参加了会议。
与会专家学者围绕生命与健康大数据开放共享、精准医学数据分析与应用、海量生物组学数据存储汇交与分析应用体系、表观遗传与生物多样性等主题进行了学术交流与讨论。国家基因组科学数据中心主任鲍一明研究员介绍了中心的成立过程、当前数据库资源和今后发展方向,以及2018年牵头成立的国际生物多样性与健康大数据联盟(BHBD)情况,得到参会人员的广泛关注和充分肯定。

第四届国际生命与健康大数据论坛
8. 建成国内领先的组学大数据存储与计算中心
随着二代测序技术在基因组研究中的广泛应用,基因组、转录组、表观组等组学数据以井喷之势爆发,生命科学研究已经进入组学大数据时代。生命与健康大数据中心,在科学院以及大型仪器设备区域共享中心(以下简称“区域中心”)的支持下,不断提高和完善所内的计算能力,已具有1Gbps总网络带宽,230万亿次/秒的聚合计算能力,配备6.5PB高性能存储系统,累计为中科院内外16家生命科研单位,87个科研机构的提供科学计算服务,平均每天活跃用户超过500人。中心用于组学数据汇交的存储系统总容量超过4PB,已建设6PB数据归档备份系统,按分级存储、安全可用的原则,通过持续提升基础设施支撑能力,实现海量组学大数据的高效管理。

中心服务器运算和存储能力增长趋势图
四、获奖及荣誉
职工
鲍一明研究员被评为2019年度北京基因组研究所身边的榜样
李茹姣高级工程师入选2019年度中国科学院关键技术人才
马利娜副研究员入选中科院2019年度“青年创新促进会会员”
组学原始数据归档库(GSA)案例被评为“2018年度中国科学院信息化优秀案例”
章张研究员被评为2018年度北京基因组研究所身边的榜样
章张研究员获得2018年中科院BHPB导师奖
郝丽丽副研究员入选中科院2018年度“青年创新促进会会员”
杜政霖高级工程师入选中科院2018年度“关键技术人才”
中心荣获2017-2018年度中央和国家机关青年文明号
赵文明高级工程师被评为2017年度北京基因组研究所身边的榜样
宋述慧副研究员入选中科院2017年度“青年创新促进会会员”
赵文明高级工程师入选中国科学院2015年度“关键技术人才”
学生
王佩获得2019年研究生国家奖学金
李兆华获得2019年研究生国家奖学金
李萌伟获得2019年研究生国家奖学金
杜强获得2019年中科院大学生奖学金
张源笙获得2019年中国科学院大学三好学生
时硕获得2019年中国科学院大学三好学生
王佩获得2019年中国科学院大学三好学生
李兆华获得2019年中国科学院大学三好学生
李兆华获得2019年优秀团员
曹佳宝获得2018年研究生国家奖学金
桑健获得2018年研究生国家奖学金
桑健获得2018年BHPB奖学金
吕洪义获得2017年研究生国家奖学金
徐行健获得2017年地奥奖学金
盛欣获得2016年研究生国家奖学金


